|
|
 |
 |
|
|
Wolfgang Marks: Die Formatierte DNA
|
|
|
Ein Netzwerk aus Antisense-Promotoren steuert die Generierung von Remodeling-Faktoren.
Zwei Netzwerke-zwei Bussysteme.
Das REMA-Gen ist durch einen antisense-Promotor und ein spezifisches NUMA-Protein definiert, das Anfang und Ende des Transkripts bestimmt. Das UsnRNA-Gen wird durch einen sense-Promotor aktiviert, der oft – aber nicht immer – mit dem antisense-Promotor des REMA-Gens identisch ist. Beide Gengruppen sind domänen-übergreifend in je einem Netzwerk organisiert, das sich an Chromosomenbändern und der Position von Promotoren in diesen Bändern orientiert.
In der IT-Welt bestehen Netzwerke im allgemeinen aus einer Vielzahl von Computern, die über die Definition von Adressräumen zu Subnetzen mit einheitlichen Zugriffs- und/oder Software-Standards oder nach anderen Regeln zusammengefasst sind. Unter den verschiedenen logischen Strukturen, die es für Computernetze gibt, ist das Bus-System schon optisch dem REMA-Netzwerk sehr ähnlich. Anstatt aus einer Vielzahl von Computern besteht das genomische REMA-Netzwerk allerdings aus ORFs bzw. Genen - aus Genen, die wie in einem Computernetz über eine bestimmte Adresse – nämlich ihren Promotor - angesprochen werden können. Diese Adressen (Promotoren) sind strangweise zu einem Bus – zu einem Adressnetz – zusammengefasst. Da jeder DNA-Strang in beiden Richtungen abgelesen werden kann, ergeben sich für das REMA-Netzwerk zwar vier Transkriptionsrichtungen, aber nur zwei Busse, da die für UsnRNAs kodierenden Gene auf dem gleichen DNA-Strang liegen wie die ORFs, an deren Spleissen zu remodeling-Faktoren sie beteiligt sind.
|
|
|
|
|
|
Meine Rasteranalysen mit den von mir berechneten Nukleosomengrössen haben ergeben, dass die Antisense und Sense-Promotoren von REMA-Genen nicht beliebig auf der DNA verteilt, sondern auf jedem DNA-Strang über eine gemeinsame einheitliche Nukleosomengrösse zu einem Netzwerk zusammengefasst sind. Den Adressraum für diese Netzwerke bildet das Chromosomenband.
Die DNA besteht aus einem gegenläufigen Doppelstrang. Auf jedem der beiden Stränge können sich Strukturgene befinden. Deshalb kommen als Zielstrang für eine Formatierung sowohl der plus-Strang als auch der minus-Strang in Betracht. Folglich muss es spezifische remodeling-Maschinen für jeden der beiden Zielstränge geben. Daraus schliesse ich: Liegen die durch eine gemeinsame Nukleosomengrösse verknüpften aktivierbaren Promotoren der REMA-Gene auf dem plus-Strang, wird eine remodeling-machine „S“ für die Formatierung definierter Chromatin-Domänen des Zielstrangs „plus“ generiert. Liegen die durch die Nukleosomengrösse verknüpften aktivierbaren Promotoren auf dem minus-Strang, wird eine remodeling-machine „C“ für die Formatierung spezifischer Domänen des Zielstrangs „minus“ gebildet.
Abhängig von der Formatierung bestimmter Abschnitte (Chromatin-Domänen) eines Chromosomenbandes können die Promotoren in diesen Abschnitten also aktivierbar sein (dann liegen sie auf der linker-DNA) oder inaktiviert sein – dann sind sie entweder an ein Histon-Oktamer gebunden oder befinden sich in einer Position relativ zum Nukleosomen-Core, die eine Aktivierung nicht zulässt.
Fassen wir zusammen: In jedem Chromosomenband und auf/in jedem DNA-Strang gibt es eine definierte Zahl (23) von Netzwerken, in welchen die sense- und antisense-Promotoren von REMA-und UsnRNA-Genen durch eine gemeinsame, einheitliche Nukleosomengrösse domänenübergreifend zusammengefasst sind. In jedem dieser Netzwerke sind die Netzwerk-Adressen (Promotor-Loci) der UsnRNA- und der REMA-Gene durch die Formatierung der jeweiligen Domänen mit einer distinkten Nukleosomengrösse derart miteinander verknüpft, dass die Promotoren, die zu einem bestimmten – durch die Nukleosomengrösse definierten - Netzwerk gehören, nur bei adresskonformer Formatierung koordiniert aktiviert werden können, da sie auch nur dann auf der linker DNA der beiden DNA-Stränge für die Transkriptionsfaktoren zugänglich sind.
Remodeling-Faktoren und die für das Spleissen dieser Faktoren benötigten UsnRNAs können also abhängig vom Zielstrang der Formatierung – abhängig davon, ob Strukturgene auf dem plus- oder dem minus-Strang exprimiert werden sollen - zum einen auf dem plus- und dem plus-reverse-, zum anderen auf dem minus- und dem minus-reverse-Strang kodiert sein. Die untenstehende Tabelle und die Grafik sollen helfen, die Verhältnisse und Beziehungen zwischen den einzelnen Faktoren des Systems deutlich zu machen.
|
|
|
|
|
REMA-Gene werden durch die Formatierung „ihrer“ Domäne mit einer bestimmten Nukleosomengrösse an- oder abgeschaltet.
Da die Aktivierung eines SP oder ASP vor allem anderen von seiner Zugänglichkeit abhängig ist – also davon, dass er in einer aktiverbaren Position auf der linker-DNA liegt – hat die unterschiedliche Formatierung einer Chromatin-Domäne auch eine unterschiedliche Zugänglichkeit der in ihr gelegenen SP von UsnRNA- und der ASP von REMA-Genen für Transkriptionsfaktoren zur Folge. Nukleosomengrössen wirken damit wie Schalter, durch die eine Gruppe von REMA- und UsnRNA-Genen an- oder abgeschaltet werden kann.
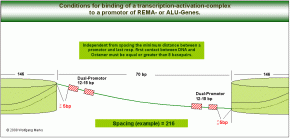 Independent from spacing the minimum distance between a promotor and last resp. first contact between DNA and Octamer incl. H1 must be equal or greater than 5 basepairs. Independent from spacing the minimum distance between a promotor and last resp. first contact between DNA and Octamer incl. H1 must be equal or greater than 5 basepairs.
Damit Transkriptionsfaktoren binden können, müssen bestimmte topologische Bedingungen erfüllt sein, die ich den Netzwerkanalysen sowohl der REMA-Gen-Promotoren als auch der ALU-Gen-Promotoren zugrundegelegt und in der Abbildung skizziert habe.88
REMA-Gene kodieren für Remodeling-Maschinen,
aber auch für Histon-Chaperone.
Die spezifische Verteilung der REMA-Gene – genauer: die Organisation ihrer Promotoren in Netzwerken, die mit distinkten Nukleosomengrössen korreliert sind - und die überaus große Zahl dieser Gene, die mit der rechnerisch erforderlichen Zahl von remodeling-Maschinen korreliert ist, lässt nur den logischen Schluss zu, dass diese Gene tatsächlich für Proteine kodieren, die in die Formatierung der DNA mit spezifischen Nukleosomengrössen involviert sind. Nach dem hier Vorgetragenen kann es kaum einen Zweifel daran geben, dass die hier erstmals beschriebenen REMA-Gene für DNA- und Histon-modifizierende Proteine kodieren, also für Remodeling-Faktoren, die sich zu remodeling-Maschinen zusammenschliessen und dann in der Lage sind, die DNA mit einer spezifischen Nukleosomengrösse (NG) zu formatieren, indem sie gezielt die Modifikationen an der DNA und den Histonen vornehmen89, die ich weiter unten unter dem Begriff „epigenetisches chromatin-remodeling“ zusammenfassen werde.
Zu den remodeling-Faktoren gehören auch spezifische ATPasen, die jedoch nicht durch REMA-Gene kodiert werden. Ihre Hydrolyse stellt die benötigte Energie für die Modifizierung der Histone und der DNA-Nukleotide zur Verfügung. Sie werden heute allgemein zur Charakterisierung und Klassifizierung der zahlreichen bisher entdeckten remodeling-Maschinen verwendet. Weitere in remodeling-Komplexen identifizierte Komponenten sind Ligasen und die sogenannten Histon-Chaperone90, die allerdings – so meine Überzeugung – durch REMA-Gene kodiert sein müssen, da ihre Tätigkeit untrennbar und unmittelbar mit der Tätigkeit der remodeling-Maschinen verknüpft ist.
Wie wird eine remodeling-Maschine gebildet?
Die reverse Transkription einer bestimmten Einheit, eines bestimmten Gens des oben gezeigten und untersuchten REMA-Gen-Clusters auf dem plus-Strang wird – wie zuvor bereits im Detail geschildert - durch einen der drei antisense-Promotoren (ASP2, ASP3 bzw. ASP5) aktiviert, der mit anderen antisense-Promotoren in der gleichen Domäne und/oder mit antisense-Promotoren in anderen Domänen (und anderen Transkriptionsgruppen) im Chromosomenband 15q22.2 zu einem Netzwerk zusammengefasst ist, dessen gemeinsames Merkmal eine einheitliche Nukleosomengrösse ist.
Ein Beispiel für ein solches domänenübergreifendes Netzwerk, das auf einer einheitlichen Nukleosomengrösse basiert, stellen die von mir analysierten REMA-Gene für die NG 232-S dar: das mit REMA-232-S/1 bezeichnete REMA-Gen auf dem plus-reverse-Strang ist mit den REMA-Genen 232-S/2 bis 232-S/4 durch die Nukleosomengrösse 204-S91 zu einem Netzwerk zusammengefasst, das nach meinen Berechnungen zusammen mit weiteren 17 REMA-Genen in anderen Domänen des gleichen Bandes remodeling-Faktoren kodiert, die sich – wie in diesem Modell gezeigt - zu einer remodeling-Maschine für die NG 232–S (TRH) zusammenschliessen.
|
|
|
|
|
Die Initiation der Transkription der in einem Netzwerk zusammengefassten REMA- und UsnRNA-Gene führt zunächst auf dem weiter oben beschriebenen Wege zur Synthese von remodeling-Faktoren und anschliessend – wie hier in einem schematischen Modell gezeigt – zur dynamischen Assemblierung einer remodeling-Maschine für die Programmierung definierter Chromatin-Domänen des Bandes 15q22.2 mit einer bestimmten Nukleosomengrösse – im gezeigten Modell mit der NG 232-S.
|
|
Zusammenfassung
Remodeling-Maschinen können höchstwahrscheinlich sowohl DNA-Nukleotide als auch Aminosäurereste von Histon-Proteinen „on the fly“ modifizieren92. Eine typische remodeling-Maschine wie die hier gezeigte müsste rein rechnerisch aus etwa 6 bis 16 verschiedenen Enzymklassen bestehen (Azetylasen, Deazetylasen, Sumoylasen, Biotinasen, Phosphorylasen, Methylasen, Demethylasen und anderen Enzymen). Insgesamt sollte das Remodelingsystem aber aus über 20 verschiedenen Enzymenklassen bestehen, die aus REMA-Gen kodierten Proteindomänen - von mir Remodeling-Faktoren (RF) genannt - nach dem „Lego-Prinzip“ zu remodeling-Maschinen assembliert werden.93
REMA-Gene werden durch die Bindung eines für den Differenzierungsschritt spezifischen Formatierungshormons an einen zellulären (kario- oder zytoplasmatischen) Rezeptor aktiviert, der danach in Untereinheiten zerfällt94. Diese aktiven Untereinheiten binden - wie zuvor detailliert beschrieben - zusammen mit anderen Transkriptionsfaktoren und der Polymerase III (U6-RNA) bzw. der Polymerase II (remodeling-Faktoren, UsnRNAs U1-U5) an hormonspezifische REMA-Gen-Promotoren in der linker-DNA von Chromatin-Domänen, die mit einer epigenetisch definierten Nukleosomengrösse formatiert sind. Ist kein Formatierungshormon vorhanden oder hat die Zelle keinen Rezeptor für ein solches Hormon ausgebildet, bindet der Transkriptionskomplex abhängig von der Art der Mitose durch die Vermittlung eines Silencing-Faktors an spezifische REMA-Gen-Promotoren für die Nukleosomengrösse 208 oder 15095.
Da die UsnRNA-Transkripte für das Spleissen der proteinogenen ORF-Transkripte benötigt werden, werden diese wahrscheinlich vor den ORFs transkribiert. Da remodeling-Faktoren und UsnRNAs (U1 bis U5) von der gleichen RNA-Polymerase transkribiert werden, dürfte die Koordination dieser beiden Prozesse für die Zelle kein unlösbares Problem darstellen. Darüberhinaus sind die Transkriptionskomplexe, die an Antisense-Promotoren binden, wahrscheinlich weitgehend identisch mit denen, die an sense-Promotoren von UsnRNAs andocken, da die Gene für die UsnRNAs und die Gene für die REMA-Faktoren sich auch oft den gleichen Promotor teilen.
Ist der Spleissvorgang abgeschlossen, werden die messenger-RNAs ins Zytoplasma transportiert und dort translatiert. Die fertigen Proteine wandern in den Zellkern und schließen sich dort zu einer remodeling-Maschine zusammen, die unter Vermittlung eines Aktivierungskomplexes (darauf komme ich noch zurück) an eine spezifische REMA-Box bindet.
Für die Aktivierung von REMA- und UsnRNA-Genen über ihre Antisense- bzw. Sense-Promotoren gelten demnach folgende Prämissen:
1. es muss ein ASP/SP-spezifisches Formatierungshormon oder ein Silencing-Faktor als Bestandteil des Transkriptionskomplexes vorhanden sein,
2. und die REMA-Gen-ASP oder UsnRNA–SP müssen für den Transkriptionskomplex zugänglich sein.
Wir können die Netzwerk-Definition von REMA-Genen also wie folgt erweitern: REMA-Gene sind in jedem Chromosomenband strangweise durch ein Netzwerk von hormonspezifischen96antisense-und sense-Promotoren zu Gruppen oder -Clustern zusammengefasst. Ihre koordinierte Aktivierung durch Transkriptionskomplexe, die entweder aktivierte Komplexe aus einem Formatierungshormon und einem Rezeptorprotein oder einen Silencing-Faktor enthalten, führt zur Transkription von ORFs, die für RNAs kodieren und von Genen für UsnRNAs, die diese RNAs zu einer messenger-RNA spleissen können. Die aus den zumeist gespleissten ORF-RNAs gebildeten mRNAs kodieren zell- und chromosomenbandspezifische remodeling-Faktoren, die sich nach der Translation nach dem Baukastenprinzip – abhängig vom kodierenden DNA-Strang - zu remodeling-Maschinen „S“ oder „C“ zusammenschließen. Die generierten remodeling-Maschinen „S“ oder „C“ modifizieren anschließend definierte DNA-Nukleotide und Aminosäurereste von Core-Histonen (Histon-Oktameren) des jeweiligen DNA-Stranges und führen so zur Programmierung einer neuen Nukleosomengrösse in allen Chromatin-Domänen dieses Bandes, die eine Bindestelle (eine REMA-Box – siehe dazu das nächste Kapitel) für die neu gebildete remodeling-Maschine aufweisen.
Remodeling-Maschinen formatieren domänenübergreifend – aber in den Grenzen von Domänen und Chromosomenbändern. Obwohl REMA-Gene - wie auch die repetitiven Elemente selbst, denen sie entstammen - über ein ganzes Chromosomenband verstreut sein können, sind die Positionen ihrer Promotoren – wie weiter oben gezeigt - nicht zufällig, sondern über definierte Nukleosomengrössen so miteinander verknüpft, dass zusammengehörende REMA-Gene auf jedem DNA-Strang gemeinsam und koordiniert über diese Promotoren aktiviert werden können. Da spezifische Rezeptoren für die von mir postulierten 19 transkriptionsrelevanten Formatierungshormone – wie in dieser Übersicht zu sehen – nur zu bestimmten Zeiten zur Verfügung stehen, ist eine koordinierte Aktivierung von REMA-Genen für die verschiedenen Nukleosomengrössen auch nur in diesen Zeitfenstern möglich. Die Expression der REMA-Gene ist also mehrfachen Kontrollen unterworfen.
Die Logik und die Einfachheit des REMA-Gen-Netzwerksystems sind ebenso bestechend wie die evolutionären Möglichkeiten gewaltig, die in ihm verborgen sind: da ein Teil LINE-Elemente noch aktiv transponierend ist, werden dem System ständig neue Bausteine, neue Proteine und RNAs hinzugefügt – es wäre also durchaus denkbar, dass der inhärente Logos des menschlichen Genoms in nicht allzu ferner Zukunft durch eine zusätzliche Nukleosomen-grösse, ein zusätzliches Formatierungshormon oder zusätzliche Möglichkeiten des alternativen Spleissens erweitert wird.
|
|
88Wie viele auch neuere Untersuchungen gezeigt haben, hängt die Aktivierbarkeit von Promotoren generell von ihrer Position auf der DNA ab. Dabei darf ein bestimmter Mindestabstand zum Oktamer nicht unterschritten werden, da sonst der Transkriptionskomplex nicht an den Promotor binden kann. Bei meinen Berechnungen zur Ermittlung der Nukleosomengrössen habe ich bestimmte Mindestabstände zwischen den Nukleosomenkernen (den Histon-Oktameren) und den Promotoren zugrundegelegt, die der Wahrheit ziemlich nahe kommen dürften.
89Während der Mitose, siehe dazu das Kapitel „Chromatin-remodeling“.
90Darüber werde ich später noch sprechen.
91Die NG204 bedeutet, dass die Chromatin-Domäne, die mit dieser NG formatiert ist, neu formatiert werden soll.
92Auch dazu werde ich später noch Ausführungen machen.
93Die Zahl der Remodeling-Faktoren entspricht vermutlich der Zahl der durchzuführenden Modifikationen an DNA und Histonen.
94Die Bildung von Hormon-Rezeptorprotein-Komplexen ist ein weit verbreitetes Prinzip der Genaktivierung in Eukarionten. Die Komplexe zerfallen in aktive Untereinheiten, die das eigentliche Agens darstellen.
95Das Silencing von Genen wird in einem gesonderten Kapitel abgehandelt.
96Ausnahme: ASP und SP von REMA-Genen, die RM für die NG 208 (markieren für Stillegung) und für die NG 150 (stillegen) kodieren
|
|
|