|
Der Weg zur zellspezifischen mRNA. Transkriptionsfaktoren: SNIRPs und TAPs.
Die Funktion der primären Transkriptionsschleife.
Durch Bindung von P6 und T2 an die Matrix/SMAR3 ist eine Doppelschleifen-Struktur entstanden - (Schmetterlingsflügel habe ich sie bei der Besprechung der REMA- und ALU-Gene genannt) – die sich aus einer inaktiven und einer aktiven DNA-Schleife zusammensetzt. Die aktive Schleife umfasst die DNA-Sequenz vom Promotor P6 bis zum poly-A-Signal T2. Sie enthält ausser den Exons 5 bis 13 eine Reihe von ALU-Genen, die für zellspezifische SNIRPs und TAPs kodieren sowie Gene für UsnRNAs, die für das Spleissen der metabolen Transkripte benötigt werden, die mit (oder aus) den Exons der aktiven Schleife gebildet werden können.
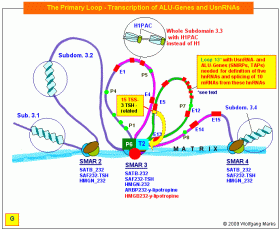 Die von mir als loop 13 bezeichnete primäre Transkriptionsschleife ist also nicht die Transkriptionsschleife, von der die Transkripte dieses Teils des TPM1-Gens generiert werden, sondern eine „Arbeitsschleife“, die ausschliesslich der Synthese von spezifischen UsnRNAs und von zellspezifischen Transkriptionsfaktoren (SNIRPs, TAPs) dient, die durch ALU-Gene kodiert sind. Die von mir als loop 13 bezeichnete primäre Transkriptionsschleife ist also nicht die Transkriptionsschleife, von der die Transkripte dieses Teils des TPM1-Gens generiert werden, sondern eine „Arbeitsschleife“, die ausschliesslich der Synthese von spezifischen UsnRNAs und von zellspezifischen Transkriptionsfaktoren (SNIRPs, TAPs) dient, die durch ALU-Gene kodiert sind.
Erst nach der Synthese dieser Faktoren und der durch diese (mit)vermittelten Bindung einer bestimmten TSS an das Transkriptosom wird die von mir sekundäre Transkriptionsschleife genannte Struktur gebildet, welche die DNA-Sequenz repräsentiert, die für die Transkription der zellspezifischen TPM1-Transkripte und deren processing massgeblich ist.
Die primäre Transkriptionsschleife 13 enthält 10 UsnRNA-Gene, deren Transkription nach dem bei den REMA-Genen beschriebenen Muster abläuft. Zudem enthält sie 10 ALU-Gene235 und die entsprechende Zahl von antisense-Promotoren. Von diesen 10 antisense-Promotoren sind 3 mit der Nukleosomengrösse 232 korreliert, liegen bei dieser Formatierung also auf der linker-DNA, aber nur einer, nämlich der ASP23, kann durch einen y-Lipotropin-HRPK in Verbindung mit je einem spezifischen SAF232-TSH bzw. ARBP232-y-Lipotropin Architekturprotein aktiviert werden. Dieser ASP23 liegt – wie in dieser Abbildung zu sehen, im 5’ upstream-Bereich zwischen dem TSS-Cluster und dem Promotor P6.
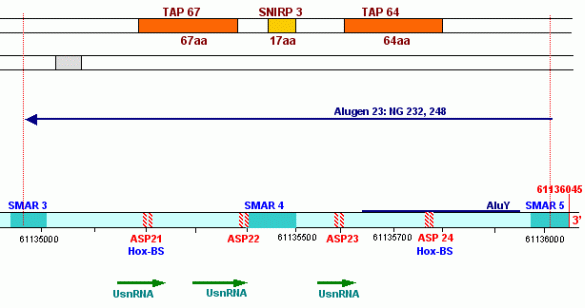 Das ALU-Gen 23 kodiert – wie auf der Abbildung zu sehen, zwei TAPs und ein SNIRP. Das TAP64 weist – wie weiter oben gezeigt, 5 Bindestellen für aktivierte HRPKs auf. Das TAP67 enthält noch einmal 5 Bindestellen (nicht gezeigt) – insgesamt verfügen die beiden durch das ALU-Gen 23 kodierten TAPs also über 10 BS für aktivierte sekundäre HRPKs. Im SNIRP3 habe ich 5 BS für aktivierte primäre HRPKs identifiziert. Ich habe ebenfalls gezeigt, dass SNIRP3 und TAP64 über die Hormonkaskade miteinander verknüpft sind und habe dies am Beispiel des SNIRP3, das unter anderem über eine TSH-HRPK BS verfügt and am Beispiel des TAP64, das ein von TSH abhängiges y-Lipotropin-HRPK binden kann, aufzuzeigen versucht. Das ALU-Gen 23 kodiert – wie auf der Abbildung zu sehen, zwei TAPs und ein SNIRP. Das TAP64 weist – wie weiter oben gezeigt, 5 Bindestellen für aktivierte HRPKs auf. Das TAP67 enthält noch einmal 5 Bindestellen (nicht gezeigt) – insgesamt verfügen die beiden durch das ALU-Gen 23 kodierten TAPs also über 10 BS für aktivierte sekundäre HRPKs. Im SNIRP3 habe ich 5 BS für aktivierte primäre HRPKs identifiziert. Ich habe ebenfalls gezeigt, dass SNIRP3 und TAP64 über die Hormonkaskade miteinander verknüpft sind und habe dies am Beispiel des SNIRP3, das unter anderem über eine TSH-HRPK BS verfügt and am Beispiel des TAP64, das ein von TSH abhängiges y-Lipotropin-HRPK binden kann, aufzuzeigen versucht.
Dass die Zahl der Bindestellen für aktivierte HRPKs im SNIRP3 und TAP64 sowie TAP67, die durch das ALU-Gen 23 in loop 13 kodiert werden, nicht zufällig 5 bzw. insgesamt 10 betragen kann, liegt auf der Hand: die 5 Bindestellen für primäre HRPKs im SNIRP3 sind über jeweils eine spezifische Nukleosomengrösse mit 5 hnRNAs korreliert, die 10 BS im TAP64 und –67 über die gleichen Nukleosomengrössen mit den 10 mRNAs, die nach meinen Berechnungen (siehe Abb.) aus diesen hnRNAs gespleisst werden können.
Das SNIRP3 mit gebundenem TSH-HRPK nenne ich von nun an das TSH-SNIRP3 – das TAP64 mit gebundenem y-Lipotropin-HRPK das Lipotropin-TAP64.
Welche Bedeutung diese beiden Transkriptions-Aktivierungsfaktoren (TAFs) haben, wird in den folgenden Kapiteln hoffentlich deutlich werden.
Der Weg zur zellspezifischen mRNA: Die Transkriptionsmaschine und ihre
Interaktionen mit der sekundären Transkriptionsschleife.
An der Bildung einer Transkriptionsmaschine sind eine Vielzahl von Faktoren beteiligt. Die im Augenblick aktuelle Modell-Vorstellung geht von etwa 40 bis 80 verschiedenen „Mitwirkenden“ aus, darunter die RNA-Polymerase II, die sogenannten allgemeinen Transkriptionsfaktoren (GTFs oder TFs) sowie spezielle Transkriptionsfaktoren (TAFs – transcription activating factors), die zell- oder gewebespezifisch sein sollen. An das Transkriptosom, das sich aus dem PIC entwickelt, sollen sogenannte Aktivatoren, Mediatoren und Repressoren direkt oder über eine DNA-Schleife herangeführt werden können. Auch die spezifischen UsnRNAs, die für das Spleissen der Exons eines bestimmten Primärtranskripts zur reifen mRNA verantwortlich zeichnen, sind oder werden nach diesem Modell Bestandteil des Transkriptosoms.
Der Core-Promotor-Komplex soll in vitro ausreichend sein zur Transkription eines chromatinfreien DNA-Stranges durch die basale Transkriptionsmaschine. Diese besteht aus der RNA-Polymerase II und den allgemeinen Transkriptionsfaktoren TFIIA, TFIIB, TFIID, TFIIE, TFIIF und TFIIH, welche jeweils (bis auf TFIIB) Multiproteinkomplexe sind (Lee und Young, 2000).
Während aktuelle Transkriptionsmodelle meines Wissens davon ausgehen, dass es nur bei der Re-Inititation der Transkription zu einer Schleifenbildung kommt, bin ich mir einigermassen sicher, dass die Schleifenbildung zwischen der TSS-Region und einem Terminator nicht die Ausnahme, sondern zumindest bei den sogenannten „Haushaltsgenen“ (household genes), zu denen auch das TPM1-Gen zählt, die Regel ist. Da es in diesem Teil meiner Arbeit aber im Wesentlichen um die Prozesse geht, die für die Definition der TSS und der poly-A-Stelle sowie für die Auswahl der Exons entscheidend sind, die in ein bestimmtes Transkript eingehen, spielt die Frage, ob die Transkriptionsschleife die Ausnahme oder die Regel ist, nur eine untergeordnete Rolle und soll an dieser Stelle nicht weiter diskutiert werden.
Im Gegensatz zu früher ist man heute allgemein der Auffassung, dass die Synthese des Primärtranskriptes236(Synonyme sind hnRNA, prä-mRNA, pre-mRNA, unreife mRNA), das sowohl Exons als auch Introns enthält und das Processing dieses Transkriptes – das Herausschneiden von Introns und das Zusammenfügen bestimmter Exons zu einer zellspezifischen mRNA – nicht zeitlich unabhängig voneinander erfolgen, sondern in einem dynamischen Prozess stattfinden, in dessen Verlauf viele der beteiligten Proteine durch Konformationsänderungen oder durch Hinzutreten neuer Faktoren neue Funktionen erlangen. Auch die Beendigung der Transkription – die Termination – und das Anfügen einer unterschiedlich langen Sequenz von Adenylatresten durch eine poly-A-Polymerase an einer definierten Stelle der RNA - der poly-A-Stelle - nach dem sogenannten poly-A-Signal ist wahrscheinlich ein Teil dieses dynamischen Prozesses.
Der Weg zur zellspezifischen mRNA:
die Bindung der Transkriptions- Startstelle an das Transkriptosom.
Damit eine zellspezifische messenger-RNA gebildet werden kann, muss der Core-Promotor und/oder das Initiator-Element an die Kernmatrix und den PIC gebunden werden. Wie und wo das geschieht und die genaue Zusammensetzung des Transkriptionskomplexes, des Transkriptosoms, ist seit vielen Jahren Gegenstand intensivster Forschung.
Waren anfangs nur die basalen Transkriptionsfaktoren bekannt, konnte das Wissen im Laufe der Jahre um eine grosse Anzahl zellspezifischer Transkriptionsfaktoren erweitert werden. Erst in neuerer Zeit wurden die sogenannten Transkriptions-Cofaktoren entdeckt, die ausschliesslich in Eukaryonten vorkommen. PC4 zum Beispiel ist ein solcher Cofaktor – er soll in Gegenwart anderer regulatorischer Faktoren die Funktion von TFIID und TFIIH unterstützen. Mediatoren schliesslich sollen eine Brücke schlagen zwischen Enhancern mit distalen regulatorischen Faktoren und der RNAP II sowie der basalen Transkriptionsmaschine. Auch die sogenannten negativen Cofaktoren sollen nicht unerwähnt bleiben – sie können, wie der negative Cofaktor 2 (NC2), die Assoziation der generellen Transkriptionsfaktoren be- oder verhindern.
Die lange allgemein vertretene Auffassung, dass die GTFs generell an den TSS-nahen Core-Promotor binden, ist – das habe ich im Kapitel Promotor und Terminator eingehend diskutiert – ins Wanken geraten. Meine eigenen Berechnungen und Untersuchungen zeigen, dass die Kopplung von Promotor P6 (heute noch als UAS oder USA bezeichnet) und Terminator T2 und die Bindung dieser beiden Motive in Verbindung mit einer SMAR der Klasse III an die Kernmatrix zum einen mit der Aktivierung und Bindung von Architekturproteinen, zum anderen mit der Rekrutierung allgemeiner und spezifischer Transkriptionsfaktoren verbunden sein muss.
Bereits bei der Besprechung der ALU-Gene weiter oben habe ich die Bindung eines hormonspezifischen ARBP- und SAF-Proteins zusammen mit den generellen Transkriptionsfaktoren TFIID und TFIIB an den Core-Promotor eines ALU-Gen-ORF als auslösendes Moment für die Bildung der sekundären Transkriptionsschleife bezeichnet.
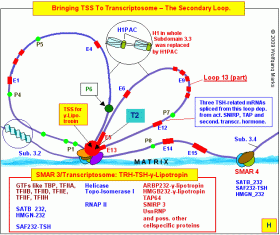 Auch beim TPM1-Gen ist die Ausbildung der sekundären Transkriptionsschleife (loop 13) an die Rekrutierung eines zellspezifischen SAF-Proteins (hier SAF232-TSH) und eines ebenso spezifischen ARBP-Proteins (hier ARBP232-y-Lipotropin), gefolgt von TFIID und TFIIB an den Core-Promotor im Exon 5 geknüpft. Dieser Komplex aus zellspezifischen Architekturproteinen, TFIIB und TFIID – in den vermutlich auch ein y-Lipotropin-HRPK integriert ist - induziert die Bindung des Core-Promotors an den Terminator T2 und gleichzeitig die Lösung des Promotors P6 aus der Bindung an die Kernmatrix/SMAR3. Dabei entsteht die in der Abbildung H gezeigte Struktur. Auch beim TPM1-Gen ist die Ausbildung der sekundären Transkriptionsschleife (loop 13) an die Rekrutierung eines zellspezifischen SAF-Proteins (hier SAF232-TSH) und eines ebenso spezifischen ARBP-Proteins (hier ARBP232-y-Lipotropin), gefolgt von TFIID und TFIIB an den Core-Promotor im Exon 5 geknüpft. Dieser Komplex aus zellspezifischen Architekturproteinen, TFIIB und TFIID – in den vermutlich auch ein y-Lipotropin-HRPK integriert ist - induziert die Bindung des Core-Promotors an den Terminator T2 und gleichzeitig die Lösung des Promotors P6 aus der Bindung an die Kernmatrix/SMAR3. Dabei entsteht die in der Abbildung H gezeigte Struktur.
Anschliessend binden TFIIE, TFIIA, TFIIF und TFIIH, die RNAPII, das TSH-SNIRP und das Lipotropin-TAP und weitere Proteine an das Transkriptosom, welche regulierende, hemmende oder verstärkende Wirkung auf die Transkription haben können.
Durch die Bindung des y-Lipotropin-Core-Promotors an das in der Matrix/SMAR verankerte Transkriptosom und die Entlassung von P6 aus der Bindung entstehen – wie in Abbildung H zu sehen - aus der primären Transkriptionsschleife wiederum zwei Schleifen, von denen die eine den nun inaktiven Teil der Subdomäne 3.3 darstellt237, die andere – die ich als sekundäre Transkriptionsschleife bezeichnet habe - jene DNA-Sequenz, aus der das zellspezifische, durch die Hormonkaskade TRH-TSH-y-Lipotropin definierte Transkript unter Mitwirkung der zuvor generierten UsnRNAs bzw. UsnRNPs (SNURPS) durch die Transkriptionsmaschine synthetisiert werden.
Die Funktion von SNIRPs und TAPs im Transkriptionsgeschehen.
An der Organisation der in Abbildung „H“ gezeigten spezifischen sekundären Transkriptionsschleife sind ausser den genannten generellen Transkriptionsfaktoren, den aufgeführten zellspezifischen Architekturproteinen und einigen (nicht aufgeführten) zellspezifischen Regulatorproteinen vor allem ALU-Gen-kodierte aktivierte SNIRPs und TAPs beteiligt, also solche, die eine aktivierte Untereinheit eines primären (SNIRP) oder sekundären (TAP) HRPKs gebunden haben238. Die wahrhaftig mannigfaltigen Beziehungen darzustellen zwischen den verschiedenen GTFs und diesen beiden Proteinklassen ist mit den Mitteln der computational analysis kaum möglich. Beide Proteine müssen aber – das ergibt sich logisch aus dem bisher Vorgetragenen, zusammen mit den hormonspezifischen Architekturproteinen an der Definition der zellspezifischen Transkripte entscheidend, wenn nicht allein bestimmend, beteiligt sein.
Aus einer Arbeit von Shaw et al.:239
„Recent experiments in yeast (Saccharomyces cerevisiae) cells have identified a species-specific region of yeast transcription factor IIB (TFIIB) located at residues 144–157. According to the human TFIIB structure, this region is part of a solvent-exposed helix in the first repeat of the carboxyl-terminal core domain. In this report, we systematically analyze four positions in this region (Lys-147, Cys-149, Lys-151, and Glu-152) that together have been shown previously to be important for yeast TFIIB’s function in vivo. Our experiments suggest that all of these four positions, and in particular positions 151, 149, and 152, are critical for yeast TFIIB’s ability to support cell growth. In addition, we describe an intragenic suppressor screening experiment to identify mutations that reverse, or partially reverse, the temperature-sensitive phenotype of a yeast TFIIB derivative bearing amino acid changes at these four positions to human residues. The suppressor mutations reveal changes at positions 115, 117, and 182 that are located outside the species-specific region of yeast TFIIB, suggesting an extended surface available to interact with other proteins. Finally, we demonstrate that the suppressor mutations restore gene activation in vivo, further supporting the idea that one important function of yeast TFIIB in living cells is to mediate gene activation.
Aus der im Ausschnitt zitierten Arbeit von Shaw et al. geht hervor, dass es sowohl im humanen TFIIB als im analogen Faktor der Bäckerhefe Sequenzen gibt, deren Mutation – experimentell herbeigeführt – zu schwerwiegenden Störungen im Wachstum der Zelle führen. Macht man die Mutationen rückgängig, ist die genaktivierende Funktion von TFIIB wiederhergestellt. Das von Shaw et al. bei der Bäckerhefe als kritisch für das Zellwachstum identifizierte Motiv umfasste die Aminosäurereste Lys-147, Cys-149, Lys-151, and Glu-152 innerhalb einer Region, die schon früher als spezies-spezifisch erkannt worden war.
Nach dem Ergebnis meiner Untersuchungen und Berechnungen sind diese spezies-spezifischen Regionen innerhalb von TFIIB Bindestellen für aktivierte SNIRPs und TAPs, also für ALU-Gen kodierte Proteine, die einen aktivierten Komplex aus einem primären bzw. einem sekundären Transkriptionshormon und einem Rezeptorprotein gebunden haben.
SNIRPs und TAPs haben – wie weiter oben gezeigt - je nach Grösse mehrere Bindestellen für unterschiedliche HRPKs – SNIRPs können insgesamt 30 verschiedene, TAPs insgesamt 52 verschiedene HRPKs binden.
Diese Zahlen sind korreliert mit dem System der Hormone, in welchem ich die gleiche Anzahl an primären und sekundären Transkriptionshormonen festgeschrieben habe. Zusammen weisen SNIRPs und TAPs also 82 BS für solche HRPKs auf - die gleiche Anzahl an Bindestellen muss deshalb auch in TFIIB zu finden sein.
 |
Diese 82 BS für aktivierte SNIRPs und TAPs, die sich grösstenteils überlappen, befinden sich etwa in einem Bereich zwischen der AS 95 und der AS 265, den ich in der Aminosäure-Sequenz des Proteins rot markiert habe. In einer gegebenen Zelle sind zu einem bestimmten Zeitpunkt natürlich immer nur (eine oder) zwei dieser BS besetzt.
Die Bindung der SNIRPs und TAPs erfolgt indirekt über die HRPKs. In der AS-Sequenz von TFIIB habe ich die BS für das aktivierte TSH-SNIRP und das aktivierte y-Lipotropin-SNIRP blau/gelb markiert.
Weiter oben habe ich mich mit der Funktion von TFIIB bei der Formierung einer Transkriptionsschleife zwischen Core-Promotor und Terminator beschäftigt und dabei auf eine Arbeit von Singh und Hampsey verwiesen240, in der sie zum Ergebnis kamen, dass die Bildung einer Schleife zwischen Promotor und Terminator zwingend von der Bindung des TFIIB an den Terminator abhängt.
Da ein poly-A-Signal mit mehreren Core-Promotoren/TSS verknüpft sein kein – bei der hier diskutierten Schleife 13 ist das auch der Fall – muss es spezifische Kriterien geben, welche die Spezifität der Kombination TSS-poly-A-Signal/Terminator definieren. Nach allem bis hierher Vorgetragenen können dies nur hormonelle Signale sein – die Bindung von aktivierten SNIRPs und TAPs an TFIIB scheint mir deshalb ein (mit)entscheidendes Kriterium zu sein für die Verbindung zwischen einer spezifischen TSS und einer bestimmten poly-A-Stelle.
Die RNA – Polymerase II und die Bedeutung der
Carboxy-terminalen-Domäne (CTD) für die Bindung von TAPs.
Dass es zwischen Core-Promotor und Terminator enge Beziehungen gibt, ist erst seit kurzer Zeit bekannt. Wurde bis weit in die 90er Jahre streng zwischen Initiation und Termination der Transkription durch Poly-Adenylierung unterschieden, setzt sich heute mehr und mehr die Auffassung durch, dass die drei wesentlichen enzymatisch kontrollierten Prozesse der Transkription – das Anfügen eines caps an das Primärtranskript, das Spleissen dieses Transkripts und die Polyadenylierung der reifen messenger-RNA in einem Maße miteinander verknüpft sind, das früher nicht für möglich gehalten wurde. Bei allen drei Prozessen kommt der sogenannten CTD – der C-terminalen Domäne der grössten von 12 Untereinheiten der RNAPII eine besondere Bedeutung zu (Überblick in: Hirose and Manley, 2000; Bentley, 2002; Maniatis and Reed, 2002).
Die RNA-Polymerase II (RNAPII) katalysiert die Bildung der meisten eukaryontischen mRNAs und snRNAs (also auch der UsnRNAs, die den Spleissprozess katalysieren) und entspricht somit formal der RNA-Polymerase der Prokaryonten, die allerdings auch die tRNA und die rRNAs transkribieren kann. Die RNA-Polymerase-Komplex der Hefe hat eine molekulare Masse von ca. 550.000 Dalton und interagiert mit zahlreichen Transkriptionsfaktoren, von denen die oben aufgeführten generellen TFs, TAFs wie SNIRPs und TAPs oder die Mediatoren nur ein Teil sind. Die RNAPII – 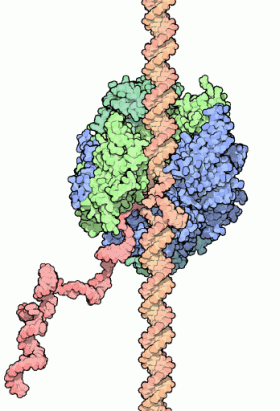 die Abbildung aus Wikipedia zeigt ein Kalottenmodell des Enzyms – hat eine Zangen- oder auch Klauenstruktur – durch die beiden Arme der Zange läuft die DNA – aus einer seitlichen Öffnung tritt die synthetisierte RNA aus, die unmittelbar danach mit einer cap-Struktur versehen wird, um sie vor dem Abbau durch allgegenwärtige Restriktionsenzyme zu schützen. die Abbildung aus Wikipedia zeigt ein Kalottenmodell des Enzyms – hat eine Zangen- oder auch Klauenstruktur – durch die beiden Arme der Zange läuft die DNA – aus einer seitlichen Öffnung tritt die synthetisierte RNA aus, die unmittelbar danach mit einer cap-Struktur versehen wird, um sie vor dem Abbau durch allgegenwärtige Restriktionsenzyme zu schützen.
Das charakteristische Merkmal der CTD ist ein repetitives Heptapeptid-Motiv, das aus den Aminosäuren Tyr1-Ser2-Pro3-Thr4-Ser5-Pro6-Ser7 besteht. 26 Mal wiederholt sich diese Motiv in in der CTD der RNAPII der Hefe, 52 mal in der CTD der RNAPII des Menschen.241 Die CTD ist Plattform für eine Reihe unterschiedlicher Proteine, die abhängig von der Modfikation der CTD an die Domäne binden oder sich wieder von ihr lösen. Als Schnittstelle zwischen Transkription und Prozessierung der entstehenden mRNA hat sie eine entscheidende Funktion bei der Kontrolle dieser beiden essentiellen Prozesse. Deletionen der C-terminalen Domäne, die über ein gewisses Mass hinaus gehen, sind letal, denn die C-Terminale Domäne ist für den Aufbau des Initiationskomplexes essentiell, da sie Proteine bindet, die mit dem Tata-Box-Bindeprotein und dem TFIID interagieren.
Dass die Serinreste der CTD durch spezifische Proteinkinasen phosphoryliert und wieder dephosphoryliert werden können, weiß man schon seit geraumer Zeit. Damit sind die Modifikationsmöglichkeiten aber wahrscheinlich nicht erschöpft: durch Prolyl-cis-trans-Isomerasen zum Beispiel können auch die beiden Prolinreste modifiziert werden.
Dass die Heptapeptid-Sequenz der CTD im Kombination mit spezifischen Modifikationen einen Code darstellen könnte, ist schon länger vermutet worden. Spezifische Phosphorylierungen an bestimmten Aminosäuren, so an Serine-2 und Serine-5 sollen die Rekrutierung von Transkriptions- und Processing-Faktoren koordinieren. Chapman et al.242 konnten mit Hilfe monoklonaler Antikörper nachweisen, dass Serine-7 phosphoryliert ist, wenn Gene aktiv transkribiert werden.
Nach dem Ergebnis meiner Untersuchungen stellen die Heptapeptid-Repeats in eukariontischen Zellen unter anderem Bindestellen für aktivierte TAPs dar – deren Zahl ist deshalb auch mit der Evolution der Genexpression in eukariontischen Zellen verknüpft. TAPs können Komplexe aus sekundären Transkriptionshormonen und membranären oder nuklearen Rezeptoren binden – es gibt deshalb ebenso viele verschiedene HRPKs, die an TAPs binden können, wie es sekundäre Transkriptionshormone gibt. Dass ich deren Zahl auf 52 festgelegt habe, war natürlich kein Zufall – müsste ich hier über das Transkriptionsssytem der Hefe referieren, hätte ich die Zahl der an Transkription oder besser Processing beteiligten sekundären Transkriptionshormone auf 26 festgelegt. Diese Zahl entspricht der Anzahl der Heptapeptid-Motive in der CTD der Hefe.
Dass SNIRPs und TAPs – bedingt durch ihre Verknüpfung mit primären bzw. sekundären Transkriptionshormonen spezifische Funktionen einmal bei der Definition des Primärtranskripts, zum andern bei der Definition der zu spleissenden Exons haben, ist nach dem hier Vorgetragenen evident. Aber auch die spezifischen UsnRNAs, die in der primären Transkriptionsschleife kodiert sind, haben wahrscheinlich Anteil an der Auswahl der Exons, die zu einer zellspezifischen mRNA gespleisst werden. Ihre Positionierung in der primären Transkriptionsschleife ist sicher nicht zufällig. Zufällig sind sicher auch nicht die arithmetischen Relationen zwischen Core-Promotor und TSS, ebenso wenig wie die zwischen TSS und DPE oder die zwischen poly-A-Signal/Terminator und poly-Adenylierungs-Stelle. Auch die Länge des sogenannten poly-A-Schwanzes, die Zahl der an die mRNA angehefteten Adenyl-Reste dürfte beim processing des Primärtranskripts/hnRNA eine Rolle spielen. Diese Relationen werde ich vielleicht zu einem späteren Zeitpunkt untersuchen.
|