|
|
 |
 |
|
|
Wolfgang Marks: Die Formatierte DNA
|
|
|
Chromatin-Remodeling – eine kritische Auseinandersetzung.
Prätranskriptionell oder posttranslational- das ist hier die Frage.
Histon- und DNA-Modifikationen sowie Histon-Varianten sind das zur Zeit wohl am intensivsten erforschte Gebiet der Biologie. Es vergeht keine Woche, in der nicht neue Entdeckungen auf diesem weiten Feld verkündet werden. Anders aber als bei einem Puzzle, bei dem mit der wachsenden Zahl von positionierten Teilen das Bild klarer und klarer in Erscheinung tritt, scheint bei den genannten Themen eher das Gegenteil der Fall zu sein.
Dies liegt sicher zum einen daran, dass die Zahl der – wie ich gesagt habe - gekoppelten Modifikationen an DNA und Histonen sehr hoch ist – in meiner Hochrechnung bin ich von 260 Modifikationen ausgegangen – zum anderen aber sicher auch daran, dass die bewussten Modifikationen bisher immer nur unter dem solitären Aspekt der Transkriptionsaktivierung gesehen und untersucht wurden. Dass der Transkription ein Prozess vorausgehen könnte – die Formatierung nämlich -, der eben die Chromatin-Strukturen etabliert, die eine zellspezifische, differenzierte Genexpression erst möglich machen, darauf scheint bis heute noch niemand gekommen zu sein. Dabei läge es doch nahe anzunehmen, dass sich die Abläufe während eines proliferativen Zellzyklus von denen eines differentiellen grundsätzlich unterscheiden, dass es also einen grossen Unterschied macht, ob die Zelle die Chromatin-Struktur nur kopiert - oder ob sie das Chromatin neu strukturiert und arrangiert, um den Zugriff auf andere Promotoren, andere Terminatoren und Transkriptionsfaktoren möglich zu machen.
Bis zur Veröffentlichung dieser Arbeit war es nicht gelungen, für die zahlreichen bis dato bekannten Histon- und Chromatin-Modifikationen, für Histon-Varianten und die verschiedenen remodeling-Komplexe und -Proteine, die an der Etablierung und an der Strukturierung des Chromatins beteiligt sind, eine integrierte Modellvorstellung zu entwickeln, die einen halbwegs logischen und verständlichen Zugang zu ihren Funktionen erlaubt hätte. Auch diese Arbeit vermag nicht alle Phänomene zu erklären, nicht alle Widersprüche aufzulösen (die sich vor allem aus in vitro –Experimenten ergeben haben). Aber sie liefert – meine ich in aller Bescheidenheit sagen zu dürfen - ein zwingend logisches und in vielen Details nachprüfbares Bild von den Prozessen, welche die differentielle Genexpression und damit die Ontogenese eines komplexen menschlichen Organismus steuern.
Chromatin-remodeling ist nicht gleich chromatin-remodeling.
Dass bestimmte chemische Modifikationen der Histon-Proteine und der Nukleotide der DNA direkte Auswirkungen auf die Transkription und die Struktur der DNA haben, wird heute von niemandem mehr bestritten. VanHolde hat bereits 1988 zeigen können, dass Histone das Ziel verschiedener posttranslationaler Modifizierungen sind, wie zum Beipiel Acetylierung, Methylierung, Phosphorylierung, Ubiquitinisierung. Heute weiß man, dass es weitere Enzyme (ADP-Ribosyl-Transferasen, Sumoylasen, Biotinasen u.a.) gibt, die Histone und auch DNA modifizieren können. Ein kleiner Teil der Enzyme/Proteine, die diese chemischen Veränderungen bewirken, ist bekannt. Auch hat man in den letzten Jahren eine Reihe von Histon- und DNA-modifizierenden Proteinen identifiziert, die sich zu Komplexen zusammenschließen, die als Remodeling-Komplexe oder „remodeling-machines“ des Chromatins apostrophiert werden. „Remodeling-Maschinen“ sollen Prozesse katalysieren, die zu einer veränderten Anordnung oder zu einer Repositionierung von Nukleosomen auf der bzw. an der DNA führen. SWR1, SWI/SNF, ISWI, BAF/BRM, ACF, RSC und NURF sind zum Beispiel solche Komplexe. Wurden diese Komplexe lange Zeit ausschließlich im Zusammenhang mit der Transkriptionsaktivierung oder der Aufrechterhaltung der Transkription und der Bildung des Prä-Initiationskomplexes gesehen123, werden Sie heute vereinzelt mehr und mehr auch mit der epigenetischen Prägung des Genoms in Verbindung gebracht.
Eines der Hauptprobleme im wissenschaftlichen Diskurs über das Thema „Chromatin-remodeling“ scheint mir zu sein, dass der Begriff chromatin-remodeling nicht genau definiert ist. So wird – vor allem in der englischsprachigen Literatur – der Begriff sowohl für die durch Histon-Acetyltransferasen, Histon-Deacetylasen, Histon- und DNA-Methyltransferasen oder –demethylasen vorgenommenen oder bewirkten covalenten Modifikationen an Histonen und DNA gebraucht, als auch auf Prozesse angewendet, die mit der Repositionierung von Nukleosomen auf der DNA einhergehen.
Um etwas Klarheit in die Materie zu bringen, schlage ich deshalb vor, grundsätzlich zu unterscheiden zwischen Veränderungen des Chromatins, die eine Repositionierung der Nukleosomenkerne (der Oktamere) relativ zur DNA beinhalten (das wäre dann „echtes“ chromatin-remodeling) und solchen Veränderungen, die zwar auch die Histone des Oktamers (und die assoziierten Nukleotide der DNA) betreffen – also das sogenannte Core -, aber nicht zu einer Verschiebung oder Neupositionierung der Histon-Oktamere auf der DNA führen und daher den Abstand von Nukleosomenkern zu Nukleosomenkern, das spacing, nicht verändern.124 Diese Prozesse sollte man besser unter den Begriff „Core-Modifikationen“ subsumieren.
Protein-Aggregate wie der Swi/Snf, ACF, ISWI, Snf2H, Chd1, Mi-2 und Brg1-Komplex sind nach dieser Definition wahrscheinlich „echte“ remodeling-Komplexe oder Untereinheiten von solchen, während die in der Literatur immer wieder zitierten und oben erwähnten HATs und HDACs, sowie die analogen HMTs, HDMTs und andere Enzyme dieser Klasse125 mit ziemlicher Sicherheit zu einer der von mir weiter unten definierten Gruppen 2, 3.1 oder 4 gehören – zumindest dann, wenn sie nicht, wie die von mir charakterisierten remodeling-Faktoren durch REMA-Gene kodiert und durch Antisense-Promotoren aktiviert werden und über eine ATP-Untereinheit verfügen (das sind die entscheidenden Kriterien), sondern durch poly- oder monocystronische „reguläre“ Gene kodiert und durch sense-Promotoren aktiviert werden.
Die von HATs, HDACs., HMTs und anderen Enzymen dieser Klasse durchgeführten Modifikationen können deshalb auch nicht zu einer Repositionierung von Oktameren relativ zur DNA126 führen, sondern lediglich zur Herabsetzung der Bindungskräfte zwischen definierten Histonen und zwischen diesen Histonen und der DNA – im englischen priming genannt - und ermöglichen dadurch eine gerichtete Deassemblierung oder auch Dissoziation definierter Histon-Moleküle von der DNA und damit die Transkription eines der beiden Stränge der zuvor in einer differentiellen Mitose formatierten DNA.
Eine Armee von remodeling-machines ist ständig mit der Neuordnung
des Chromatins beschäftigt.
Zur Zeit werden Remodeling-Komplexe als ATP-abhängige Multiproteinkomplexe charakterisiert, deren Masse zwischen 0,5 und 2 MDa liegen soll. Allen gemeinsam ist eine ATPase-Untereinheit, die auch zur Kennzeichnung der einzelnen Komplexe herangezogen wird.
Kannte man bis Ende 2006 nur eine Handvoll solcher remodeling-Komplexe, wächst die Zahl der identifizierten Protein-Aggregate zur Zeit rasant. Bis heute (August 2008) hat man ca. 40 remodeling-Komplexe entdeckt – eine Übersicht zeigen K. Rippe et al. in ihrer 2007 veröffentlichten Arbeit „DNA sequence- and conformation-directed positioning of nucleosomes by chromatin-remodeling complexes.126a”
|
|
|
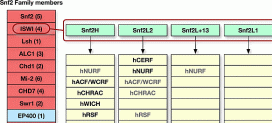 Abbildung links mit freundlicher Genehmigung aus K. Rippe et al. (2007): „DNA sequence- and conformation-directed positioning of nucleosomes by chromatin-remodeling complexes.” Abbildung links mit freundlicher Genehmigung aus K. Rippe et al. (2007): „DNA sequence- and conformation-directed positioning of nucleosomes by chromatin-remodeling complexes.”
|
|
Rippe et al. schätzen, dass es in allen menschlichen Zellen zusammengerechnet einige hundert bis über tausend verschiedene remodeling-Komplexe geben muss. Damit stimmen die Schätzungen der Autoren größenordnungsmässig gut mit meinen Berechnungen am Schluss des nächsten Kapitels “Der differentielle Zellzyklus” überein.
In der zitierten Arbeit wird unter anderem gezeigt, dass die von remodeling-Komplexen (hier ACF, ISWI, Snf2H, Chd1, Mi-2 und Brg1) bewirkte Positionierung von Nukleosomen auf der DNA nicht zufällig, sondern an bestimmten hotspots erfolgt, die – unter anderem - durch die DNA-Sequenz an diesen Stellen prädestiniert sind. Andere Untersucher hatten auch früher schon belegen können, dass Histon-Oktamere im Hefe-Genom zum Beispiel einmal so positioniert werden, dass bestimmte Promotoren inaktiviert werden – ein andermal aber so, dass Bindestellen für Transkriptionsfaktoren auf der linker DNA für diese Faktoren zugänglich werden.
Rippe et al. schließen aus ihren Versuchen, dass remodeling-Maschinen offensichtlich keine simplen Nukleosomen-„Beweger“ sind, sondern das Chromatin auf eine ganz bestimmte Art und Weise organisieren.
Zitat:
„In light of these findings and the results reported here, the high abundance and diversity of remodeling complexes suggests that chromatin-remodeling machines are specific chromatin organizers and not simply nucleosome mobilizers. Their activity seems to be directed by two additional information layers encoded in the DNA sequence. One would represent binding preferences of the histone octamer to certain DNA sequence elements. It has been shown for yeast that about half of the in vivo nucleosome positions can be predicted solely from the underlying DNA sequence. These sites are likely to provide thermodynamically favorable histone–DNA interactions, and the data reported here suggest that they also are selected as preferred locations in the remodeling reaction. However, the relative occupancy of these sites can be strongly affected by the chromatin-remodeling complexes. The coupling of their specific activity with intrinsic nucleosome preferences for certain DNA sequences could contribute significantly to determining nucleosome locations in living cells. Accordingly, the targeting of a significant fraction of nucleosomes to their DNA sites in the cell cannot be predicted without including the characteristic activities of chromatin-remodeling complexes present at the respective genomic loci. This view is consistent with a recent analysis of nucleosome locations in yeast that points to the involvement of additional factors in the determination of nucleosome positions. As demonstrated here, one important parameter to be considered is the binding affinity of the remodeler and the nucleosome. Either because of a sequence-specificity of remodeler–DNA interactions or more indirectly by an altered nucleosome structure, a reduced remodeler–nucleosome interaction leads to the release of nucleosomes to these sites. Thus, the positioning of nucleosomes in the cell could involve a chromatin-remodeling code127. Features encoded by the DNA sequence are recognized by chromatin-remodeling complexes to establish specific nucleosome-positioning patterns that define the accessibility of DNA and with it on or off states for DNA-dependent processes.“
Diese Organisation hängt nach den Ergebnissen der zitierten und (auch anderer) Arbeiten zum einen von der Sequenz der DNA ab, zum anderen von den spezifischen Eigenschaften - oder besser - Fähigkeiten der remodeling-Komplexe.
Histon-Varianten erhöhen die Spezifität des remodelings.
Erst in neuerer Zeit128 hat man entdeckt, dass es remodeling-Maschinen gibt, die offensichtlich in der Lage sind, die Zusammensetzung der Nukleosomen-Cores noch nach dem replikativen Zusammenbau (also außerhalb der S-Phase) der Oktamere zu verändern, indem sie den Austausch eines Standard-Histons – im konkreten Fall H2A – gegen eine Histon-Variante – hier H2A.Z - katalysieren. Der SWR1-Komplex aus Saccharomyces cerevisiae, für den dies nachgewiesen wurde, soll aus 13 verschiedenen Untereinheiten bestehen – als eine dieser Untereinheiten konnten Krogan et al. (2003) und Kobor et al. (2004) den bereits bekannten Bromodomainfactor 1 (Bdf1) identifizieren. Bdf1 enthält zwei sogenannte Bromodomänen, von denen man weiß, dass sie spezifisch mit azetylierten Histonen interagieren.
Auch Wu WH et al. 129 haben die Beziehungen zwischen bestimmten Untereinheiten des SWR1-Komplexes und H2A.Z genauer untersucht und herausgefunden, dass ein Teil der Untereinheiten für den Transport, andere für die Bindung von H2A.Z an das Nukleosom notwendig sind. Und schließlich ist es laut Wu et al. die carboxy-terminale alpha-Helix von H2A.Z, die der Erkennung durch SWR1 dient.
Bruno et al. (2003) haben in einem invitro–Modell-System die Bedingungen für den Austausch von kompletten H2A-H2B-Dimeren durch ATP-abhängige remodeling-Komplexe untersucht und zeigen können, dass nur Komplexe aus der SWI/SNF-Familie dazu in der Lage sind, Komplexe der ISWI-Familie dagegen nicht.
Es wird immer offensichtlicher, dass der Austausch von Histon-Varianten gegen canonische Histone nicht auf einzelne Histone wie H2A oder H3 beschränkt ist, sondern auch andere Core-Histone unmittelbar oder mittelbar einschließt. Der Austausch eines canonischen Dimers gegen eine paraloge dimere Variante könnte zwar ein singuläres Ereignis sein, welches als solches die Dissoziation und Reassemblierung des ganzen Oktamers auslöst, wie manche Autoren vermuten (Boeger et. al. 2003; Reinke and Hörz, 2003) – es könnte aber auch – und das ist für mich viel wahrscheinlicher und sehr viel logischer – Teil einer konzertierten remodeling-Aktion sein, die den simultanen Austausch nicht nur eines Histon-Dimers, sondern den Austausch von zwei oder mehr canonischen Dimeren (oder auch von canonischem H1) gegen paraloge Varianten zum Ziel hat.
An dieser Stelle erhebt sich natürlich die Frage, welchen Sinn, welche Funktion der Austausch von canonischen Histonen gegen Histon-Varianten hat. Offensichtlich gibt es für den Austausch spezifischer canonischer Histone gegen spezifische paraloge Varianten auch spezifische remodeling-Komplexe, wie Mizuguchi et al. (2004) in einem in vitro – System beobachtet haben: sie konnten nachweisen, dass SWR1 – unabhängig vom Replikationsgeschehen - in der Lage ist, heterogene H2A.Z-H2B-Dimere gegen homogene H2A-H2B-Dimere auszutauschen. Diese Reaktion war ATPase–abhängig und auf den SWR1-Komplex beschränkt. Darüber hinaus konnten Mizuguchi et al. zeigen, dass die beschriebene katalytische Funktion sich ausschließlich auf H2A.Z erstreckt und nicht auch auf canonisches H2A.
Es liegt auf der Hand, dass der Austausch von canonischen Histonen gegen Histon-Varianten wahrscheinlich durch die gleichen REMA-Gen-kodierten remodeling-Komplexe mit ATPase-Untereinheit erfolgt, wie die anschließende oder simultane Modifizierung der beiden Histon-Klassen. Was noch fehlt, ist die Logik, die diese beiden Prozesse miteinander verbindet.
|
|
|
Chromatin-Remodeling:
Versuch einer neuen Ordnung
Bevor ich im Detail auf einzelne Aspekte des chromatin-remodelings und seine Bedeutung für den Zellzyklus, die Mitose und die Differenzierung einer Zelle eingehe, will ich versuchen, das Thema „chromatin-remodeling“ mit neuer Systematik und neuer Nomenklatur neu zu strukturieren.
Chromatin-remodeling findet statt
1. als konservatives remodeling bei der Replikation der DNA als Vorbereitung zur Zellteilung
2. als selektives remodeling prä- bzw. cotranskriptionell als Vorbereitung zur Transkription eines der beiden DNA-Stränge (priming)
3. als epigenetisches remodeling unterteilt in zwei Phasen
3.1 als normatives remodeling (markieren der geplanten Veränderungen) - Vorbereitung zur Neuformatierung
3.2 als institutionelles remodeling - Neuformatierung der DNA - als Basis der differentiellen Genexpression
4. als konstitutives remodeling im Zuge der Chromosomenkondensation
Von den unter 1. bis 3.2 aufgeführten remodeling-Prozessen sind meines Wissens bisher nur die Prozesse 1 und 2 teilweise beschrieben. Das unter Punkt 4 genannte konstitutive remodeling – die Verpackung der DNA in mehreren Stufen - ist nur in Ansätzen verstanden – ich werde darauf in Teil III dieser Arbeit zurückkommen. Dass der von mir selektives remodeling genannte Prozess keine epigenetische Funktion hat, sondern lediglich – wie oben schon angedeutet - der Vorbereitung der Transkription eines der beiden DNA-Stränge dient, kann ich im Moment zwar nicht belegen, wird aber später aus dem Zusammenhang heraus deutlich werden. Gleiches gilt für das normative remodeling, das den ersten Schritt des epigenetischen remodelings darstellt.
Beschäftigen werde ich mich also zunächst mit den Punkten 1 bis 3.
1. Konservatives chromatin-remodeling
Für die Vorgänge bei der Restrukturierung des Chromatins während der DNA-Replikation in einer proliferierenden Zelle gibt es gute Modelle, die der Wahrheit wahrscheinlich ziemlich nahe kommen. Man geht davon aus, dass die Nukleosomen während der Replikation „gesplittet“ oder deassembliert werden: Die Untersuchung der Verhältnisse in einem biochemisch gut zugänglichen Test-System, dem natürlichen "Minichromosom" Simian Virus 40 (SV40) lässt den Schluss zu, dass die Struktur der Histon-Oktamere vor der Replikationsgabel teilweise aufgelöst und hinter der Replikationsgabel reorganisiert wird. Dabei spielt die inhärente Struktur des Histon-Oktamers wahrscheinlich die entscheidende Rolle.
Erinnern wir uns: Die Histonklassen H2A/H2B auf der einen, H3 und H4 auf der anderen Seite können unabhängig voneinander Aggregate bilden. Bei Assoziationsversuchen aggregieren die argininreichen Histone H3 und H4 zum Tetramer 2xH3/2xH4, die schwach lysinreichen Histone bilden zunächst ein Dimer und anschließend ebenfalls das Tetramer 2xH2A/2xH2B. Das Oktamer besitzt also eine a priori "zweigeteilte" oder besser „viergeteilte“ Struktur, die das splitting bzw. das Disassemblieren der Tetramere und Dimere – abhängig von spezifischen Histon – Modifikationen – in definierten Ebenen prädestiniert.
Neuere Studien, auf die ich später noch zurückkomme, zeigen allerdings, dass die H3- und H4-Histone während der Replikation wahrscheinlich nicht, wie bisher angenommen, als Tetramere, sondern analog zu ihren H2A-H2B-Pendants als H3-H4-Dimere auf die Tochterstränge verteilt werden.
Konstitutives chromatin-remodeling gibt die nukleosomalen Muster - das Genomformat - der Mutterzelle auf die Tochterzellen weiter: die Expressionsmuster von Mutter- und Tochterzellen sind nach der Teilung also identisch.
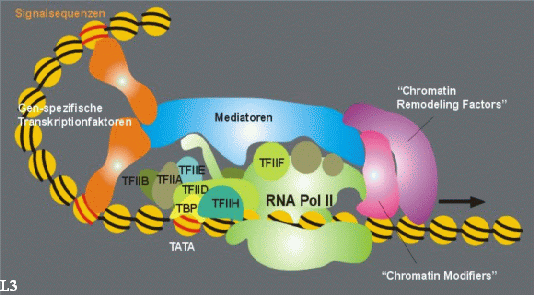
Soweit sich Replikation (Abb. L1) und DNA-Synthese im Kontext von Zell-Proliferation abspielen, will ich es mit dieser (sehr) kurzen Übersicht bewenden lassen. Wer mehr über die aktuellen Modell zum Ablauf der Replikation und der Transkription wissen will, findet auf den Webseiten der Ludwig-Maximilians-Universität München weiterführende Informationen. Die folgenden drei Abbildungen wurden mit freundlicher Genehmigung diesen Seiten130 entnommen.
|
|
|
|
|
|
 |
2. Selektives prätranskriptionelles remodeling
Die Verhältnisse bei der Transkriptionsaktivierung haben offensichtlich große Ähnlichkeit mit den Vorgängen bei der Replikation der DNA: Auch bei der Initiation der Transkription muss die DNA im Bereich des Promoters und/oder des Enhancers zugänglich sein. Die zur Zeit herrschende Vorstellung vom Ablauf der Aktivierung und der Elongation der Transkription zeigen die Abbildungen L2 und L3.
DNA, die um eine Histon-Oktamer gewickelt ist, kann nicht transkribiert werden. Soweit sind sich alle Untersucher einig. Ob die Nukleosomen-Cores aber ganz oder teilweise aufgelöst werden, ob und welche Histone an der DNA bleiben und in welchem zellulären Kontext das geschieht, darüber gehen die Meinungen auseinander. Das im Augenblick favorisierte Modell der Transkriptionsaktivierung geht von einem initialen Komplex aus, der aus allgemeinen auf der einen, gen- oder zellspezifischen Transkriptionsfaktoren auf der anderen Seite sowie aus remodeling Maschinen, Mediatoren (Chaperonen) und der Polymerase II selbst besteht. Anders ausgedrückt: differentielle Genexpression wird interpretiert als ad hoc-Veränderung der Nukleosomenkonfiguration eines genomischen locus, speziell der Promotor-Region. Es scheint ein allgemeiner Konsens dahingehend zu bestehen, dass chromatin-remodeling-Komplexe prätranskriptional aktiv sind. Welcher der beiden DNA-Stränge abgelesen, transkribiert wird, soll von der Struktur des Promotors abhängen. Welche Strukturen eines humanen Promotors zur Transkription des einen oder des anderen Stranges führen sollen, darüber ist meines Wissens nichts bekannt.
Nach meiner festen Überzeugung sind für die Auswahl des zu transkribierenden Stranges in einer gegebenen Zelle nicht der Promotor (jedenfalls nicht der Promotor des zu transkribierenden Strukturgens) sondern die spezifischen Modifikationen verantwortlich, die ein definierter ATP-abhängiger Remodeling-Komplex an der DNA und den Histonen in einer vorhergehenden Phase der Zellentwicklung – in einer vorhergehenden differentiellen Mitose - vorgenommen hat. Von diesen strangspezifischen Modifizierungen hängt es in erster Linie ab, wie die Nukleosomen-Cores deassemblieren, welche Histone aneinander und an die DNA gebunden bleiben und welche von ihr dissoziieren.
Wie ich in den einleitenden Bemerkungen zu diesem Kapitel bereits gesagt habe, sollten wir in Zukunft grundsätzlich unterscheiden zwischen solchen Histon- (und DNA)-Modifikationen, die zur Repositionierung von Histon-Oktameren auf der DNA, also zu einem veränderten spacing führen und solchen, die lediglich die Bindungskräfte zwischen einzelnen Komponenten des Nukleosomen-Cores oder die Bindungskräfte der Cores untereinander modifizieren, die Bindung der Histone an die DNA aber nicht tangieren. Die histon-modifizierenden Enzyme, die während der Transkription tätig werden, werden dies auf der Basis der während der Mitose vorgenommenen Formatierung. Sie geben zwar die Fahrt für die remodeling-Maschinen frei – um in einem Bild zu sprechen – die Weichen allerdings wurden lange vor Fahrtbeginn gestellt.
Die histon-modifizierenden Enzyme, die spezifischen Histondeazetylasen, -demethylasen, -dephosphorylasen und Ubiquitinasen, die während der Transkription für die kontrollierte Deassemblierung der Histone und ihre gerichtete Dissoziation sorgen, verändern die Anordnung der Nukleosomen-Cores auf der DNA also nicht.
Die Chromatin-remodeling–Komplexe wie der ACF oder ISWI dagegen tun dies sehr wohl – sie agieren aber nicht in einem gemeinsamen Zeitfenster mit dem Transkriptionskomplex, sondern verrichten ihre Arbeit transkriptionsunabhängig während ganz bestimmter Phasen der Mitose. Das werde ich später noch im Detail erläutern.
In diesem Zusammenhang scheinen mir die Beobachtungen einer Forschungsgruppe um J. Griesenbeck an der Universität Regensburg erwähnenswert: man hat dort die Korrelation zwischen spezifischen Histon-Modifikationen und der Genaktivität untersucht und unter anderem belegen können, dass die Aktivierung eines RNA Polymerase II-abhängigen eukaryotischen Gens mit dem Verlust von nukleosomalen Strukturen am Promotor einhergeht. J. Griesenbeck und seine Gruppe konnten auch belegen, dass die Nukleosomen durch einen Disassemblierungs-Mechanismus aufgelöst werden.131
Interessant ist auch eine Beobachtung im Zusammenhang mit FACT, einem Komplex, von dem bisher nur bekannt war, dass er an der Elongation der Transkription beteiligt ist: FACT katalysiert nach den Beobachtungen von Belotserkovskaya et al. (2003) während der Transkription sowohl die Entfernung von H2A-H2B-Dimeren aus dem Oktamer als auch die Reassemblierung derselben. Damit verhält sich FACT ähnlich wie Spt6, das schon einige Zeit als Histon-Chaperon bekannt war und von Kaplan et al. (2003) als Komplex identifiziert wurde, der ebenfalls in der Lage ist, Chromatin-Strukturen wiederherzustellen, die im Zuge der Polymerase II-Elongation aufgelöst wurden.
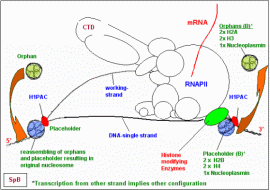 Ich hatte mich bereits 1990/91 mit dem Problem auseinandergesetzt, wie eine DNA, die auch während der Transkription offensichtlich (elektronenoptisch manifes- tiert) mit Nukleosomen besetzt ist, transkribiert werden kann und hatte meine Vorstellungen vom Ablauf der Transkrip- tion wie hier gezeigt (Originalzeichnung aus meiner Arbeit von 1990 ) skizziert. Ich hatte mich bereits 1990/91 mit dem Problem auseinandergesetzt, wie eine DNA, die auch während der Transkription offensichtlich (elektronenoptisch manifes- tiert) mit Nukleosomen besetzt ist, transkribiert werden kann und hatte meine Vorstellungen vom Ablauf der Transkrip- tion wie hier gezeigt (Originalzeichnung aus meiner Arbeit von 1990 ) skizziert.
Wie unschwer zu erkennen ist, bin ich damals bereits davon ausgegangen, dass die Struktur der Nukleosomen-Cores, der Oktamere, zum Zweck der Transkription zumindest teilweise nach einem bestimmten Muster aufgelöst wird. Abhängig von der hormonellen Initiation der Transkription sollte es – so meine damalige Vorstellung - zur Bildung von verschieden konfigurierten „Ersatz- oder „Platzhalter-Nukleosomen“ kommen, die zum einen Teil aus den originären Histonen des Oktamers bestehen sollten, zum anderen Teil aus gebundenem Nukleoplasmin und Untereinheiten von aktivierten Hormon-Rezeptor-Proteinkomplexen (HRPKs). Nach dem Durchgang der Transkriptionsmaschine sollten diese Ersatz-Nukleosome wieder durch die originären Histone ersetzt werden. Nach meinem Wissenstand von heute war dieses historische 1990er-Modell im Großen und Ganzen nicht so sehr falsch. Wie ich den Ablauf dieses Prozesses heute sehe, werde ich in Teil III dieser Arbeit schildern.
Die bisher bekannten und propagierten Chromatin-remodeling-Modelle beruhen also – ich hatte das am Anfang dieses Kapitels schon erwähnt – auf einigen wenigen bekannten Modifikationen an der DNA und an bestimmten Histonen, die entweder mit der Transkriptionsaktivierung oder dem Silencing, der Inaktivierung von Genen in Verbindung gebracht werden. Ein wie auch immer geartetes Muster – ein System – liess sich aus den bisher bekannten Modifikationen und ihrer funktionalen Interpretation aber nicht ableiten.
3. Epigenetisches remodeling oder die Verwirklichung
des epigenetischen Programms.
Eine der Hauptschwierigkeiten im Verständnis von DNA- und Histonmodifikationen scheint mir in der Interpretation der Begriffe „posttranslational“ bzw. „prätranskriptionell“ zu liegen. Wie ich hoffe deutlich gemacht zu haben, sind auch posttranslationale Modifikationen von Histonen letztlich dann prätranskriptionell, wenn der Sinn oder das Ziel dieser Modifikationen im Focus des Untersuchers liegen. Wenn Histone posttranslational von remodeling-Maschinen modifiziert werden - woran wohl kaum ein Zweifel besteht – müsste das durch diese bewirkte Chromatin-remodeling der Transkription der involvierten Gene mehr oder weniger unmittelbar vorausgehen. Aus mehreren Gründen, von denen ich einige weiter oben schon genannt habe, dürfte es für die Zelle schwierig sein, diese verschiedenen Prozesse – Transkription der Histone, Transkription der remodeling-Maschinen, Modifikation der Histone, Bestückung der DNA mit den modifizierten Histonen, Aktivierung und Regulation der Transkription von metabolischen Genen – in einem einzigen mitotischen Zellzyklus unterzubringen. Das von mir hier vorgestellte neue Remodeling-Modell und Zellzyklus-Modell geht deshalb von der Prämisse aus, dass die einzelnen Stadien der Differenzierung einer Zelle sich auf einen prämitotischen und einen postmitotischen Zellzyklus verteilen, von denen der zweite Zellzyklus unvollstängig bleibt (wenn man Mitose mit Zellteilung gleichsetzt), weil die Zelle in diesem zweiten Zellzyklus nach dem Durchlaufen einer Dekondensations- und einer remodeling-Phase unmittelbar in die Arbeitsphase übergeht.
Das zur Zeit propagierte Remodeling-Modell macht auch meines Wissens weder eine Aussage darüber, ob und wie die beschriebenen Histon-Modifikationen mit der Auswahl eines poly-A-Signals bzw. einer poly-A-Stelle verknüpft sind, noch darüber, wie über die Auswahl einer TATA-Box oder eines TATA-Box-adäquaten Promotors verschiedene Primärtranskripte (hnRNAs, prä-mRNAs) definiert werden und auch keine Aussage darüber, ob und wie das chromatin-Remodeling mit dem Processing eines Primärtranskripts zu verschiedenen messenger-RNAs verbunden ist.
Ich hoffe in dieser Arbeit gezeigt zu haben, dass es neben dem zuvor beschriebenen selektiven prä- oder cotranskriptionellen chromatin-remodeling ein epigenetisches remodeling gibt, das dem prätranskriptionellen remodeling vorausgeht und unabhängig von Transkription oder Nicht-Transkription eines bestimmten Gens erfolgt.
|
|
|
123Es wird aber zunehmend deutlicher, dass ein hoher Acetylierungsgrad in Promotor-Bereichen in vielen Fällen nicht direkt mit dem Ausmaß an Gentranskription korreliert ist, sondern einen Zustand „voraktivierten Chromatins“ darstellt, welches unter anderem auch einen potentiell aktivierbaren Promotor beinhalten kann. Dieser Zusammenhang wird im Englischen auch als „Potentiation of Chromatin“ oder „Priming“ bezeichnet (O'Neill and Turner 1995; Ford et al. 1996; Kramer et al. 1998; Francastel et al. 2000)
124Wie weiter oben schon gesagt, definiere ich das Nukleosom als Einheit aus Nukleosomenkern (Oktamer mit 146 bp DNA) und linker-DNA bis zum ersten DNA-Kontakt des nächsten Oktamers.
125Zu dieser Gruppe gehören auch die sogenanten Sirtuine, Histon-Deacetylasen der Klasse III, die in jüngster Zeit unter anderem mit der Regulierung von Alterungsvorgängen, der Stress-Resistenz und der Transkription in Verbindung gebracht werden.
126Man liest auch in neueren Arbeiten immer wieder, dass Histon-Oktamere in Promotor-Bereichen durch die enzymatische Aktiviät von Remodeling-Komplexen auf der DNA „verschoben“ werden können. Ich hoffe deutlich gemacht zu haben, dass sich die Neupositionierung von Nukleosomen nicht auf Promotor-Bereiche beschränkt, also kein lokales Phänomen ist, sondern größere DNA-Bereiche Chromatin-Domänen) umfasst und den ersten Schritt der Genaktivierung darstellt. Neuere Untersuchungen weisen auch darauf hin, dass neben lokal eng umgrenzten Regionen (z.B. in Core-Promotorbereichen) auch sehr viel größere Bereiche ständig globalen Acetylierungs- und Deacetylierungsprozessen unterworfen werden (Vogelauer et al. 2000).
126a http://www.pnas.org/content/104/40/15635.abstract
127Hervorhebung durch den Autor
128Krogan et al., 2003; Mizuguchi et al., 2004; Kobor et al., 2004
129Wu WH, Alami S, Luk E, Wu CH, Sen S, Mizuguchi G, Wei D, Wu C (2005) Swc2 is a widely conserved H2A.Z-binding module essential for ATP-dependent histone exchange. Nat Struct Mol Biol 12(12):1064-71
130http://biochemie.web.med.uni-muenchen.de/biotutor_2004/polII_transcription.htm#Modulation
131Boeger, H., Griesenbeck, J., Strattan, J.S. and Kornberg, R.D. (2003): Nucleosomes unfold completely at a transcriptionally active promoter. Mol Cell, 11, 1587-1598
|
|
|