|
|
 |
 |
|
|
Wolfgang Marks: Die Formatierte DNA
|
|
 |
|
Der Weg zur Transkriptionsschleife:
das Netzwerk der ALU-Gene.
Die ALU-Gene-Teil I.
Es gilt mittlerweile als gesichert, dass Transkription nicht „irgendwo im Zytoplasma“, sondern kernmatrixassoziiert in definierten Chromosomenkompartimenten stattfindet. Die Bindung der SMARs an die Kernmatrix wird durch Proteine bewirkt, von denen ich die vermutlich entscheidenden in dieser Arbeit aufgezählt und besprochen habe.
Damit ein bestimmtes Gen transkribiert werden kann, müssen an definierten Stellen der DNA (SMARs, Promotoren, Terminatoren, TSS) komplexe Transkriptions- und Spleissmaschinen installiert oder aktiviert werden, in denen ausser den von mir oben aufgeführten Architekturproteinen eine große Zahl weiterer Faktoren in dynamischem Zusammenwirken aktiv sind. Dazu gehören vor allem die RNA-Polymerasen II und III, aber auch Topo-Isomerasen, Helikasen und die sogenannten allgemeinen und spezifischen Transkriptionsfaktoren.
Allgemeine Transkriptionsfaktoren wirken per definitionem zellunabhängig an Transkriptionsprozessen mit - spezifische Transkriptionsfaktoren dagegen werden nur in einer bestimmten Zelle aktiviert. Während die allgemeinen Transkriptions- oder Initiationsfaktoren TFIIA, TFIIB, TFIID, TFIIE, TFIIF und TFIIH, welche jeweils (bis auf TFIIB) Multiproteinkomplexe darstellen (Lee und Young, 2000) bekannt und relativ gut untersucht sind, sind die spezifischen Transkriptionsfaktoren, Kofaktoren und Mediatoren nur zum Teil bekannt - und selbst die Rolle der bekannten Kofaktoren bei der Bildung und der Positionierung des Transkriptosoms ist weitgehend unverstanden. Insbesondere die Herkunft, der Ursprung der zell- und entwicklungsspezifischen Transkriptionsfaktoren und die Wege zu ihrer Aktivierung liegen grösstenteils im Dunkeln.
Bevor ich am Beispiel des TPM1-Gens versuche zu zeigen, welche Prozesse und Faktoren an der Aktivierung der Transkription, an der Definition einer Transkriptionsschleife, einer hnRNA und einer messenger-RNA beteiligt sind, will ich zuvor über eben diese zell- und entwicklungsspezifischen Transkriptionsfaktoren sprechen.
Der Autor dieses Textes ist sich sicher, dass es neben den von ihm entdeckten REMA-Genen ein zweites analoges Gen-System gibt, das einen essentiellen, wenn nicht den entscheidenden Teil der zell- und entwicklungsspezifischen Transkriptions- und Spleissfaktoren kodiert.
Dieses bereits mehrfach erwähnte Gen-System – von mir ALU-Gene genannt – entspricht in seinem logischen Aufbau und in der Art der Anordnung der kodierenden Sequenzen weitgehend dem zuvor in Teil II beschriebenen REMA-Gen-System. Alles, was ich dort über die Organisation des REMA-Gen-Netzwerks gesagt habe, trifft im Prinzip auch auf ALU-Gene, ihre Organisation, Aktivierung und Transkription zu. Mit zwei Ausnahmen allerdings:
ALU-Gene werden nicht durch Formatierungshormone, sondern durch primäre Transkriptionshormone aktiviert und sie sind nicht auf der Ebene von Chromosomenbändern, sondern auf der Ebene von Subdomänen organisiert.
An dieser Stelle sei mir erlaubt, einen Passus zu zitieren, den ich an den Beginn des Teil II dieser Arbeit gestellt habe:
Beiden von mir entdeckten und hier erstmals beschriebenen Gensystemen ist eines gemeinsam: sie wären ohne einen inhärenten logos, ohne das zuvor postulierte Nukleosomengrössen-System nicht denkbar. Denn sowohl die REMA-Gene als auch die ALU-Gene bilden Netzwerke, in denen die Promotoren dieser Gene durch eine einheitliche Nukleosomengrösse zusammengefasst sind. Erst die Organisation der Promotoren der REMA- und ALU-Gene in solchen Netzwerken macht die koordinierte Aktivierung von Promotoren und somit die koordinierte Transkription und Translation der beteiligten remodeling-Faktoren (REMA-Gene) beziehungsweise Transkriptions- faktoren (ALU-Gene) möglich.190
Über Alu-Sequenzen wird seit kurzem intensiv geforscht. Allerdings gehen alle Voruntersucher, soweit ich die Quellen einsehen konnte, bei ihren Bemühungen, einen Sinn in den in grosser Zahl über das Genom verstreuten repetitiven Sequenzen zu finden, von „sense“-Transkripten aus, also von Transkripten vom plus- oder vom minus-Strang jeweils in der 5’ ==>3’ –Richtung.
Ausserdem konzentriert sich die Forschung auf AluJ-, AluS- und AluY-Elemente, die evolutionär jüngsten Vertreter dieser Gruppe repetitiver Elemente.
Die entscheidenden, proteinkodierenden Sequenzen der ALU-Gene, die ORFs, welche die zellspezifischen Transkriptionsfaktoren kodieren, die ich SNIRPs (Small Nuclear Interspersed Repetitive Protein) bzw. TAPs (TAFIIB assoziiertes Protein) genannt habe, verbergen sich aber ähnlich wie die remodeling-Faktoren der REMA-Gene in antisense-Transkripten des plus- und des minus-Stranges. Und ähnlich wie bei den REMA-Genen finden sich auch in den ALU-Gen-Clustern Gene für UsnRNAs, die an Spleissprozessen beteiligt sind. Allerdings spleissen diese UsnRNAs nicht die Transkripte der proteinogen ALU-Gen-ORFs, sondern die Transkripte von metabolen Genen, die sich in der gleichen Subdomäne befinden wie sie selbst. Darauf komme ich später noch zurück.
Die entsprechenden DNA-Abschnitte enthalten zwar oft – aber nicht immer – auch ein Alu-Element aus der AluJ-, AluS- oder AluY-Familie - die proteinkodierenden Sequenzen, die ich ALU-Gene genannt habe, entstammen jedoch nicht den AluJ-, AluS- oder AluY-Elementen (dazu sind diese viel zu jung), sondern deren Vorläufern. Die Suche nach dem Ursprung dieser Vorläufer wiederum führt uns viele hundert Millionen Jahre, wahrscheinlich fast 3 Milliarden Jahre in eine Zeit zurück, in der sich aus einem Ur-7SL-RNA-Gen das erste fossile Alu-Monomer entwickelt hat.
Auf den folgenden Seiten soll deshalb versucht werden zu klären, wie die Familie der ALU-Gene entstanden ist – oder entstanden sein könnte - und auf welche Weise und in welchem Umfang antisense-Transkripte von ALU-Genen an der Aktivierung von Genen, der Regulation der Transkription und am Spleissprozess selbst beteiligt sind.
ALU-Gene: lebendige Zeugen der zellulären Evolution
Aus „wikipedia – die freie online-Enzyklopädie:
„Alu-Sequenzen sind eine Familie repetitiver (sich wiederholender) DNA-Sequenzen in Genomen von Primaten. Sie gehören zu den "Short interspersed nucleotide elements" (kurze, verteilte Nukleotidelemente), abgekürzt SINEs. Alu-Sequenzen sind jeweils circa 300 Basenpaare (bp) lang, machen jedoch über 10% des menschlichen Genoms aus (das entspricht einer Kopienanzahl von über 1 Million). Sie sind besonders häufig in den überdurchschnittlich genreichen R-Banden zu finden. Alu-Sequenzen
werden durch die RNA-Polymerase III transkribiert, jedoch nicht translatiert, es werden also RNAs gebildet, diese jedoch nicht in Proteine übersetzt. 191
Die Sequenz wurde 1978 von Catherine M. Houck und Kollegen im Menschen entdeckt. Sie wurde nach dem Restriktionsenzym Alu I (aus Arthrobacter luteus) benannt, weil es diesen Abschnitt in zwei Teile auftrennt. Es entsteht ein 170 bp und ein 130 bp-Element.“ (Zitat Ende)
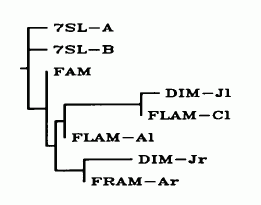 Yves Quentin hat sich bereits 1992 mit dem Ursprung der Alu-Familie befasst192 und alle Alu-Elemente auf zwei 7SL-RNA Vorläufer-Sequenzen zurückgeführt, die er als 7SL-A und 7SL-B bezeichnet hat (Abbildung: Yves Quentin). Aus diesen entwickelte sich die FAM-Familie (FAM für Fossil Alu Monomer) und aus diesen wiederum die FLAM- (Free Left Alu Monomer) und die FRAM-Gruppe (Free Right Alu Monomer). Durch Fusion des FLAM und des FRAM-Elements entstand laut Quentin schliesslich das uns heute bekannte Alu-Dimer-Element. Yves Quentin hat sich bereits 1992 mit dem Ursprung der Alu-Familie befasst192 und alle Alu-Elemente auf zwei 7SL-RNA Vorläufer-Sequenzen zurückgeführt, die er als 7SL-A und 7SL-B bezeichnet hat (Abbildung: Yves Quentin). Aus diesen entwickelte sich die FAM-Familie (FAM für Fossil Alu Monomer) und aus diesen wiederum die FLAM- (Free Left Alu Monomer) und die FRAM-Gruppe (Free Right Alu Monomer). Durch Fusion des FLAM und des FRAM-Elements entstand laut Quentin schliesslich das uns heute bekannte Alu-Dimer-Element.
Nach Quentin lassen sich drei Phasen in der Entwicklung der Alu-Elemente unterscheiden: in der ersten Phase entsteht aus einem 7SL RNA-Ur-Gen durch Deletion einer 141 bp-Sequenz zunächst das fossile Alu-Monomer (FAM). In einem zweiten Schritt entwickelt sich aus diesem durch Deletion einer 42bp-Sequenz das freie linke Alu-Monomer (FLAM) und später wiederum aus der ursprünglichen FAM-Sequenz durch Deletion einer 11 bp-Nukleotidfolge das freie rechte Alu-Monomer (FRAM). Durch die Fusion dieser beiden Elemente und Aufnahme einer 31 bp-Sequenz, deren Bedeutung noch unbekannt ist, entsteht schließlich das uns bekannte Alu-Element.
In der Phase, in der sich das FRAM und das FLAM-Element bildeten – so vermutet Quentin – könnten die ersten Alu-Monomere einen neuen Promotor193 entwickelt haben, der sie in die Lage versetzte, der transkriptionellen Regulation der 7SL RNA-Gene zu entkommen und von da an eigene Wege zu gehen.
Die zuvor erwähnten Alu-Elemente AluJ-, AluS- und AluY treten zum ersten Mal vor ca. 100 Millionen Jahren auf – und zwar nur bei den Mammaliern. Man teilt sie nach ihrem evolutionären Alter in 3 Gruppen ein und diese wiederum in Untergruppen: Als älteste Elemente mit einem Alter von ca. 60 bis 100 Mill. Jahren gelten die AluJ-Elemente, darauf folgen mit einem Alter von ca. 25 bis 60 Mill. Jahren die AluS- und darauf wiederum mit einem Alter von bis zu 30 Mill. Jahren die AluY-Elemente. AluJ und AluS machen etwa 83% aller Alu-repeats aus, nur etwa 17% sind der jüngsten und einzig noch aktive194 Gruppe der AluY-Elemente zugehörig (Roy-Engel et al., 2001).
AluJ-, AluS- und AluY-Elemente stehen aus verschiedenen Gründen seit geraumer Zeit im Focus der Forschung. Zum einen wird schon seit längerer Zeit vermutet, dass Alu-Elemente - weil sie in genreichen Abschnitten der Chromosomen besonders häufig sind - in welcher Form auch immer mit der Expression von Genen verknüpft sind. Dies ist an der untenstehenden Abbildung sehr gut zu sehen: sie zeigt Metaphase-Chromosomen eines menschlichen Lymphozyten - durch Fluoreszenz-in-situ-Hybridisierung (FISH) wurden die Alu-Sequenzen grün markiert. Die DNA ist rot eingefärbt, dadurch werden auch die gen-armen Regionen sichtbar. |
|
|
|
|
Abbildung mit freundlicher Genehmigung entnommen aus: Bolzer A, Kreth G, Solovei I, Koehler D, Saracoglu K, et al. (2005) Three-Dimensional Maps of All Chromosomes in Human Male Fibroblast Nuclei and Prometaphase Rosettes. PLoS Biol 3(5): e157 doi:10.1371/journal.pbio.0030157.
|
|
|
Zum anderen können retrotransponierte Alu-Elemente oder solche, die durch homologe Rekombination weitergegeben wurden, zur Ursache von Krankheiten werden. Auf diese Art in „fremde“ Genom-Bereiche inserierte Alu-Elemente sollen u.a. die Hämophilie B, Hypercholesterinämie und die Duchenne-Muskeldystrophie auslösen (Deininger u. Batzer, 1999). Beobachtet wurde auch, dass Zellen unter Stress vermehrt Transkripte von Alu-Sequenzen generieren – dieses Phänomen hat offensichtlich auch einen direkten Einfluss auf die Translation von messenger-RNA (Chu et al., 1998). In diesem Zusammenhang solll nicht unerwähnt bleiben, dass auch eine andere Klasse von repeats – die sogenannten Mikrosatelliten – mit bestimmten Krankheiten wie z.B. der Chorea Huntington assoziiert werden: diese tandemartig wiederholten Sequenzen aus 2 bis ca. 10 Nukleotiden sind über das gesamte Genom verstreut und machen immerhin etwa 0,5% des Genoms aus. Dies ist m.E. ein deutlicher Hinweis darauf, dass für die korrekte Funktion genetischer Elemente die arithmetische Ordnung innnerhalb der funktionellen Abschnitte der DNA eine wichtige Rolle spielt.
Weil die Entwicklung der Alu-Elemente eng mit der Evolution der Primaten verbunden ist, hat sich die aktuelle Forschung vor allem auf die jüngeren Alu-Elemente konzentriert. Dabei ist offensichtlich übersehen worden, dass die Entwicklung der Mechanismen, welche die Expression von Genen in einer eukariotischen Zelle steuern, in eine Zeit zurückreichen muss, in der von diesen Elementen allenfalls Vorläufer existierten. Grundlegende, auch heute noch vorhandene Regelwerke der Zelle führen uns sogar in die RNA-Welt der Ur-Zellen zurück – in eine Welt, in der die RNA der Träger der Erbinformation war.
|
|
ALU-Gene stammen von den Progenitoren der Alu-Elemente, also vom freien rechten (FRAM) und vom freien linken Alu-Monomer (FLAM) ab.
Die Prozesse, über die wir hier sprechen, liegen viele hundert Millionen Jahre zurück. Der folgende Exkurs kann deshalb naturgemäss nur ein Versuch sein, sich den tatsächlichen Abläufen anzunähern.
Wahrscheinlich begann das Leben auf der Erde vor ca. 4 Milliarden Jahren mit der Bildung sogenannter Mikrosphären, die der Biochemiker Sidney Fox bereits Mitte der 1950er Jahre im Labor herzustellen in der Lage war. In verschiedenen Versuchen gelang es ihm nachzuweisen, dass sich Aminosäuren unter hohem Druck und großer Hitze zu kleinen Hohlkügelchen von ca. zwei Tausendstel Millimeter Durchmesser arrangieren, die durch Kontakt mit Wasser zu wachsen beginnen und schon so etwas wie einen Stoffwechsel aufweisen, indem sie selektiv bestimmte Stoffe aus der Umgebung aufnehmen oder solche Stoffe ausscheiden. Diese Mikrosphären waren vermutlich die ersten echten Protozellen. Viele Millionen Jahre später, etwa 3,8 Milliarden Jahre vor unserer Zeit, hatten sich bereits kernlose RNA-haltige Zellen entwickelt, die manchen unserer heutigen Bakterien ähnlich sind. Rund achthundert Millionen Jahre später, vor ca. 3 Milliarden Jahren, wurde dann wahrscheinlich der Vorläufer der ersten Hormone „erfunden“ – die Arachidonsäure. (Die Arachidonsäure-Abkömmlinge oder Eicosanoide (Prostaglandine, Leukotriene, Thromboxane) allerdings entstanden erst viel später, vor ca. 2 Milliarden Jahren).
Vor etwa 3 Milliarden Jahren geschah etwas sehr einschneidendes: einem frühen Retrovirus gelang die Integration eines Teils seiner RNA ins zelluläre (RNA)-Genom195 . Damit gelangten retrovirale Enzyme, ein 7-SL-RNA-Vorläufer und ein Antisense-Promotor in die Zelle sowie der erste, noch inkompetente Teil196 eines Mechanismus, den wir heute als Retrotransposition kennen.
Es dauerte aber noch einmal etwa 500 Millionen Jahre, bis sich vor etwa 2,5 Milliarden Jahren die ersten eukariotischen RNA-Urzellen bildeten, die mit einem Zellkern und einer Zellmembran ausgestattet waren, wie sie die heutigen Zellen im Prinzip auch noch besitzen. Diese Veränderung ging Hand in Hand mit einer Veränderung der Atmosphäre: bedingt wahrscheinlich durch die Abkühlung des Erdmantels und den dadurch ausgelösten Rückgang von Vulkanausbrüchen, die mit der Lava den für die methanbildenden Bakterien lebenswichtigen Nickel in den Urozean gespült hatten, fehlte den bis dahin das Leben im Wasser dominierenden methanbildenden Bakterien dieser für sie lebenswichtige Rohstoff. Die Folge: die sauerstoffbildenden Algen und Cyano-Bakterien gewannen die Oberhand und reicherten die Atmosphäre von nun an mehr und mehr mit Sauerstoff an197.
Vor rund 2 Milliarden Jahren muss sich dann das erste fossile Alu-Monomer FAM gebildet haben. Aus dem antisense –Promotor entwickelte sich ein zusätzlicher sense-Promotor, mit dessen Hilfe FAM der Regulation durch die Transkriptionsmaschinerie der 7SL-RNA-Gene entkommen und sich von da an mit Hilfe der retroviralen Enzyme über das Genom der Zelle verbreiten konnte. Auch das System der Nukleosomengrössen und das Verpackungssystem der DNA muss in dieser Zeit des grossen Umbruchs „erfunden“ worden sein ebenso wie ein rudimentäres (autokatalytisches?) Spleiss-System und die ersten UsnRNAs.
Es dauert aber noch einmal viele (ca. 400) Millionen Jahre, bis vor ca. 1,8 bis 1,7 Milliarden Jahren ein oder auch mehrere einschneidende Ereignisse eintraten, die letztlich erst die Entwicklung auch höheren pflanzlichen und tierischen (mehrzelligen) Lebens möglich machten: die Integration bakterieller (Teil)genome in die Zelle.
Nach der heute allgemein anerkannten Endosymbionten-Theorie sind wesentliche Zellsysteme der Eukarionten nämlich durch (unvollständige) Endozytose von konkurrierenden Zellformen entstanden. Sowohl die Chloroplasten der Pflanzen, als auch die Mitochondrien der Tiere (und Pilze) sollen nach dieser Theorie aus endozytotisch inkorporierten Bakterien stammen. Dieses Ereignis hatte aber ausser der Hinzugewinnung wichtiger Zellorganellen noch eine weitere Folge: das „Ur“-Line1-Element und mit diesem verknüpfte funktionelle genetische Elemente wurden in das Zellgenom integriert. So entstand durch die Kombination (retro)viraler und bakterieller Reproduktionssysteme das Konstrukt Retrotransposition.198 Retrotransposition sorgte von da an sowohl für die Verbreitung der LINE1-Elemente, aus denen sich das System der REMA-Gene und der remodeling-Maschinen entwickelte, als auch der FAM-Elemente, aus denen FLAM und FRAM und damit die ALU-Gene entstanden, welche die von mir weiter oben erwähnten zellspezifischen Transkriptionsfaktoren kodieren.
Beide Gensysteme bedienen sich also bis zum heutigen Tag der antisense- und sense-Promotoren, die aus der viele hundert Millionen Jahre zurückliegenden Integration eines viralen Teilgenoms in das Genom der Zelle stammen.199
Etwa um diese Zeit auch – 1,5 bis 1,2 Milliarden Jahre vor unserer Zeit – dürften das erste free left Alu-monomer FLAM und die Vorläufer der HMG-Proteine in einer schon recht weit entwickelten Zelle das „Licht der Welt erblickt“ haben. FLAM besass den gleichen dualen antisense/sense -Promotor wie FAM und bediente sich für seine Verbreitung deshalb auch der gleichen Retrotranspositions-Maschinerie wie sein Vorläufer FAM.
Von diesem freien linken Alu-Monomer FLAM stammt mit grosser Wahrscheinlichkeit ein (sehr) kleines Protein ab, nämlich ein weiter unten beschriebener zellspezischer Transkriptionsfaktor, den ich „SNIRP“ genannt habe.
Der Erwerb der neuen Zell-Organellen durch symbiotische Endozytose, vor allem die Inkorporation der Mitochondrien als „Kraftwerke“ der neuen Zellen markiert zugleich den „Startschuss“ für die Entwicklung mehrzelliger Tiere und Pflanzen: allerdings dauerte es noch einmal einige hundert Millionen Jahre, bis diese Entwicklung tatsächlich realisiert werden konnte. Denn erst durch den Übergang von der RNA als Informationsträger und –speicher zur DNA als Träger der Erbinformation – der vor ca. 1,1 Milliarden Jahren stattfand - wurden die Voraussetzungen für die Entwicklung komplexerer Lebensformen, für die Entwicklung mehrzelliger Tiere und Pflanzen geschaffen. Da durch einen auf vier Symbolen beruhenden genetischen Code mehr Informationen gespeichert werden konnten als durch einen Code, der auf lediglich drei Codons beruhte, hatten Zellen, die DNA als Informationsspeicher nutzten, den RNA-Zellen gegenüber einen entscheidenden evolutionären Vorteil.
Zur gleichen Zeit – also vor etwa 1,1 Milliarden Jahren – entwickelte sich aus FAM das freie linke Alu-Monomer FRAM, auf das ein zweiter wichtiger Transkriptionsfaktor zurückgeht: ein ebenfalls relativ kleines Protein, das ich – siehe oben - TAP (TAFIIB associated Protein) genannt habe, weil es mit dem sogenannten allgemeinen Transkriptionsfaktor TAFIIB korreliert ist.
Hand in Hand mit dem Übergang von der RNA zur DNA vollzog sich die Differenzierung in immer komplexere und vielzelligere Lebensformen: vor rund einer Milliarde Jahren begann sich an Land eine erste Flora aus Urfarnen und im Wasser eine Fauna aus Schwämmen und primitiven Weichtieren zu entwickeln200.
Die ersten nachweisbaren fossilen Lebensspuren datiert man zur Zeit auf ca. 750 Millionen Jahre vor unserer Zeit. Vom Kambrium an schliesslich ist die Entwicklung des tierischen Lebens durch Fossilien halbwegs gut dokumentiert (weil die verschiedenen Lebensformen Hartschalen und Panzer auszubilden begannen) und kann in jedem Lehrbuch nachgelesen werden.
Die Entwicklung der ALU-Gen-Progenitoren allerdings war damit noch nicht beendet: denn mit der Fusion von FRAM und FLAM vor rund 100 Millionen Jahren begann zeitgleich mit der Evolution der verschiedenen Alu-Familien auch die der Plazentatiere und schliesslich vor ca. 45 Millionen Jahren die der Primaten. Der Homo sapiens „sapiens“ dürfte sich vor etwa 15 Millionen Jahren von seinen äffischen Urahnen getrennt haben und versucht seit dieser Zeit wenig erfolgreich der Bezeichnung „sapiens“ gerecht zu werden.
|
|
|
190Diese Aussage trifft im Grunde auf alle Gene, gleich welcher Klasse oder Art zu. Die metabolen Gene zum Beispiel sind in Netzwerken organisiert, die aus Domänen, Subdomänen und Promotoren bestehen. Ich werde das weiter unten in diesem Kapitel noch am Beispiel des TPM1-Gens zeigen.
191Dies trifft für antisense-Transkripte nicht zu. Diese werden auch translatiert.
192Origin of the Alu family: a family of Alu-like monomers gave birth to the left and the right arms of the Alu elements; Yves Quentin; Nucleic Acids Research, 1992, Vol. 20, No. 13; 3397-3401
193Das geschah meines Erachtens um einiges früher. Siehe den folgenden Text.
194Die Aussage, dass im menschlichen Genom ausschließlich Alu-Y-Elemente noch aktiv retrotransponieren, mag zwar richtig sein, täuscht aber leicht über die Tatsache hinweg, dass auch oder gerade die phylogenetisch älteren interspergierten Elemente (FAM, FLAM, FRAM) in der Zelle zu funktionellen Gruppen zusammengefasst sind und in dieser Gruppierung maßgeblich an der Regulation der Genexpression beteiligt sind.
195Eine symbiotische Verbindung, welche die Entwicklung der Zelle entscheidend geprägt hat. Ohne sie gäbe es Leben in seiner jetzigen Form nicht.
196Der zweite Teil kam vor ca. 1,6 Milliarden Jahren durch symbiotische Endozytose eines Bakteriums hinzu. Dies ist eine These, die meines Wissens hier zum ersten Mal aufgestellt wird.
197Oceanic nickel depletion and a methanogen famine before the Great Oxidation Event; Kurt O. Konhauser et al.; Nature 458, 750-753 (9 April 2009)
198Da der Mechanismus der Retrotransposition – für dessen Entstehung ich hier erstmals eine logische Erklärung liefere - sich bis heute erhalten hat, muss dieser der Zelle einen gravierenden evolutionären Vorteil verschafft haben.Umso erstaunlicher ist es, dass man am Max-Planck-Institut für Entwicklungsbiologie erst jetzt die Auswirkung der Retrotransposition auf die Evolution des Menschen aufzuklären bemüht ist. (Elena Khazina, Oliver Weichenrieder: Non-LTR retrotransposons encode noncanonical RRM domains in their first open-reading frame. PNAS, 20. Januar 2009, vol. 106 (3), 731-736)
199Die beiden wichtigsten Gensysteme der eukariotischen Zelle entspringen also einer symbiotischen Beziehung zu einem Retrovirus bzw. einem oder mehreren Bakterien.
200Ich postuliere demnach, dass die ersten pflanzlichen und tierischen Lebensformen vor rund einer Milliarde Jahren entstanden, also in einer Zeit, die weit vor der in Nature (Nature; 457, 718-721) genannten Zeit für das Vorkommen von Schwämmen (ca. 750 Millionen Jahre) liegt. Die sogenannte "Kambrische Explosion", in der sämtliche Tierstämme ihren ersten, plötzlichen Auftritt auf der Bühne der Evolution gehabt haben sollen, hat es in dieser Form wahrscheinlich nicht gegeben. Die „Kambrische Explosion“ ist m.E. ein artefakt, der auf artbedingte Lücken im Fossilbestand zurückgeht.
|
|
|