|
ALU-genes and UsnRNA-genes coding factors needed for expression and splicing of metabolic gene(s) within subdomain are transcribed from same strand. Antisense transcription (5’ <== 3’) generates little proteins called SNIRPs and TAPs (naming by author). Transcription in sense-direction (5’ ==> 3’) generates the well-known UsnRNAs, which are involved in processing of hnRNAs/primary transcripts derived from metabolic gene on same strand.
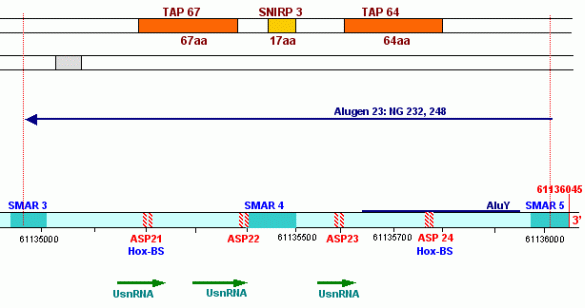
Die folgende Abbildung zeigt die Analyse der zuvor angesprochenen etwa 2000 Basenpaare umfassenden Sequenz vom Basenpaar 61134027 bis zum Basenpaar 61136045 aus dem Intron zwischen Exon 4 und Exon 5 des TPM1-Gens. Dieser Abschnitt befindet sich zwischen einem von mir definierten/berechneten203 Promotor (P6) und dem Exon 5 des genannten Gens.
Dieser von mir im Detail untersuchte DNA-Abschnitt, der von SMARs der Klasse II eingerahmt bzw. unterbrochen wird, enthält sowohl auf dem plus-Strang als auch auf dem minus-Strang je fünf antisense-Promotoren, welche die Transkription und Translation von Proteinen initiieren, die durch ORFs auf dem plus-reverse- bzw. minus-reverse Strang kodiert werden. Ausserdem werden auf dem plus-Strang in 5’ ==>3’ (sense) - Richtung drei UsnRNAs kodiert. Über die Funktion dieser UsnRNAs werde ich weiter unten noch sprechen.
Der hier gezeigte DNA-Abschnitt enthält demnach 5 ALU-Gene, die ich mit ALU-Gen 20 bis 24204bezeichnet habe. Ihre Aktivierung und (antisense-) Transkription in der Interphase durch die RNA-Polymerase II ist von der Formatierung der Domäne 3 mit einer bestimmten Nukleosomengrösse abhängig. Der ASP23 zum Beispiel kann nur aktiviert werden, wenn die Domäne 3 (und damit natürlich auch alle Subdomänen in dieser Domäne) mit der Nukleosomengrösse 232 (TRH) oder 248 (GH-RH) formatiert sind. Nur dann liegt dieser Promotor auf der linker DNA zwischen zwei Nukleosomencores.205
Die Formatierung mit der Nukleosomengrösse 232 muss dabei von einem bestimmten Startpunkt aus erfolgen, nämlich vom TSH-Startpunkt. Die Aktivierung eines ALU-Gen antisense-Promotors ist also zum einen von der Formatierung der Domäne mit einer bestimmten Nukleosomengrösse abhängig – und damit von einem Formatierungshormon und einem von diesem induzierten primären Transkriptionshormon (TRH/TSH), zum anderen aber auch von der Bindung eines Transkriptionskomplexes, in dem ein Rezeptorprotein und ein sekundäres Transkriptionshormon (HRPK) enthalten sind, das wiederum vom primären Transkriptionshormon (TSH) abhängig ist.
Die Aktivierung einer spezifischen remodeling-Maschine - und die Formatierung einer Domäne von einem bestimmten Startpunkt aus206- ist also mit der Aktivierung eines oder mehrerer spezifischer ALU-Gene – und damit mit der Generierung zellspezifischer Transkriptionsfaktoren - über die Hormonkaskade, genauer: über ein Formatierungshormon, ein primäres und ein sekundäres Transkriptionshormon logisch - heute würde man sagen logistisch -verknüpft.
ALU-Gene kodieren für zwei Klassen von Proteinen, denen
entscheidende Funktionen bei der Aktivierung und der Regulation der Genexpression zukommen: SNIRPs208 und TAPs.
Zwischen den REMA-Genen und den ALU-Genen gibt es bei aller Ähnlichkeit der Genstrukturen ausser den bereits genannten einen weiteren entscheidenden Unterschied: die proteinogenen antisense-Transkripte der REMA-Gene werden mit Hilfe von UsnRNAs gespleisst, die auf dem gleichen DNA-Strang kodiert sind. Erst durch das Spleissen der proteinogenen REMA-Gen-Transkripte wird eine translatierbare messenger-RNA erzeugt.
ALU-Gen-Transkripte dagegen werden nach dem Ergebnis meiner Analysen nicht gespleisst, sondern nach Transkription und Translation von spezifischen Restriktionsenzymen in einzelne Proteine zerlegt - das Translationsprodukt des ALU-Gens 23 zum Beispiel in je ein Protein aus 67, 64 und 17 Aminosäureresten. Die innerhalb der jeweils aktiven Transkriptionsschleife kodierten UsnRNAs werden also nicht für das Spleissen der proteinogenen ALU-Gen-Transkripte, sondern vielmehr für das Spleissen des Primärtranskripts des metabolen polycistronischen Gens in der jeweiligen Subdomäne verwendet.
Die SNIRPs.
Wie in dieser Abbildung gezeigt, ist jedes der von mir definierten ALU-Gen-Transkripte durch SMARs der Klasse II begrenzt und durch Bindestellen für NuMA-Proteine charakterisiert, die mit einem bestimmten Formatierungshormon und damit auch mit einer bestimmten Nukleosomengrösse verknüpft sind. Dabei überschneiden sich die Transkripte zum Teil. Das bedeutet, dass die Transkription eines bestimmten SNIRPS oder TAPs auch durch mehr als einen Promotor angestossen werden kann.
 Das durch das ALU-Gen23 kodierte SNIRP3 mit 17 aa weist insgesamt fünf verschiedene Bindestellen für aktiverte HRPKS aus einem primären Transkriptionshormon und einem Rezeptorprotein auf. Das durch das ALU-Gen23 kodierte SNIRP3 mit 17 aa weist insgesamt fünf verschiedene Bindestellen für aktiverte HRPKS aus einem primären Transkriptionshormon und einem Rezeptorprotein auf.
Das SNIRP 3 zum Beispiel ist Bestandteil dreier verschiedener Transkripte, nämlich der Transkripte ALU-Gen22, -23 und –24, die von den gleichnamigen Promotoren aktiviert werden, also durch die ASP22, -23 und -24. Die Aktivierung dieser Promotoren wiederum ist an die Formatierung des ALU-Gens mit bestimmten Nukleosomengrössen gebunden, im konkreten Fall sind dies die Nukleosomengrössen 232, 240, 244, 248 oder 252: Der ASP 22und das ALU-Gen 22
sind mit den NG 240 und NG 244 verknüpft.
Der ASP 23 und das ALU-Gen 23
sind mit den NG 232 und NG 248 verknüpft.
Der ASP 24 und das ALU-Gen 24
sind mit der NG 252. verknüpft.
Das Transkript des ALU-Gens 23, das bei Formatierung der Domäne 3 mit der NG 232 durch den ASP23 aktiviert wird, reicht von einem bestimmten Punkt innerhalb der SMAR 5, der durch ein NuMA232-Protein bestimmt ist, bis zu einer Bindestelle für ein homologes NuMA232-Protein innnerhalb der SMAR 3. Es kodiert 3 Polypeptide, davon zwei TAPs mit 67 bzw. 64 Aminosäuren und ein SNIRP, das aus 17 Aminosäuren besteht. Betrachten wir zunächst dieses SNIRP, seine Proteinsequenz und seine von mir postulierte Funktion etwas genauer:
 |
SNIRPs sind – wie schon erwähnt - sehr kleine Polypeptide, die nach meinen scans nur zwischen ca. 13 und ca. 33 Aminosäurereste aufweisen, vielleicht mit ein Grund dafür, warum man sie bis heute nicht entdeckt hat oder nicht beachtet hat. Sie weisen abhängig von Ihrer Grösse mehrere Bindestellen für aktivierte Komplexe (HRPK, engl.:HRPC) aus einem primären Transkriptionshormon auf, das von einem Formatierungshormon und einem Rezeptorprotein abhängig ist.
Die den Bindestellen jeweils zugeordnete Kombination von Nukleosomengrösse bzw. Formatierungshormon und primärem Transkriptionshormon korrespondiert – wie könnte es anders sein – mit den Nukleosomengrössen, den Formatierungshormonen und primären Transkriptionshormonen, welche auch die Transkription des ALU-Gens 23 aktivieren.
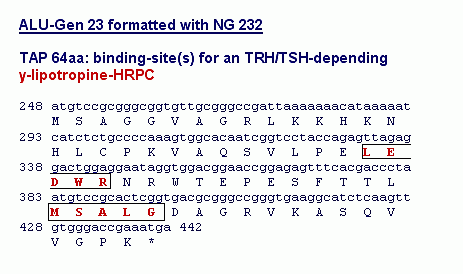
Die Abbildung zeigt das Transkript von SNIRP 3 (17aa), seine Aminosäure-Sequenz und die von mir postulierten Bindestellen für aktivierte Hormon-Rezeptorproteinkomplexe in Abhängigkeit von der Formatierung des ALU-Gens 23 mit den von mir berechneten Nukleosomengrössen.
Das SNIRP 3 kann also in verschiedenen Phasen der Ontogenese – korreliert mit jeweils unterschiedlicher Formatierung der Domäne 3 – unterschiedliche Komplexe aus einem von 30 primären Transkriptionshormonen und einem korrelierten Rezeptorprotein binden. SNIRPS, die einen solchen HRPK gebunden haben, nenne ich aktivierte SNIRPs. Welche Funktion diese aktivierten SNIRPs haben und wie sie mit der Definition einer hnRNA – mit der Definition des eines bestimmten Promotors und eines bestimmten Terminators verknüpft sind, werde ich nach der Vorstellung der TAPs diskutieren.
Die TAPs.
Die beiden durch das ALU-Gen 23 kodierten TAPs weisen 64 bzw. 67 Aminosäurereste auf. Sie sind also wesentlich grösser als die SNIRPs. TAPs können nach meinen scans zwischen ca. 45 und ca. 105 Aminosäurereste aufweisen. Wie in Abbildung XX zu sehen ist, sind bestimmte SNIRPs und TAPs zu Transkriptions- und damit Funktionseinheiten zusammengefasst. Im ALU-Gen23 bilden sowohl das TAP 67aa als auch das TAP 64aa mit dem SNIRP 3 (17aa) eine solche Einheit.
Ich werde mich hier ausschliesslich mit dem 64aa-Protein beschäftigen, da es mit dem SNIRP3 über die Nukleosomengrösse 232 bzw. das Formatierungshormon TRH und das primäre Transkriptionshormon TSH verknüpft ist. TRH und TSH spielen neben dem SNIRP3 und dem TAP64 bei der Expression einer spezifischen TPM1-hnRNA eine wichtige Rolle: während das SNIRP3 an der Definition dieser hnRNA beteiligt ist, ist das TAP 64 in das processing/splicing derselben hnRNA zu einer spezifischen mRNA involviert.
Wer meine Arbeit bis hierher aufmerksam gelesen hat, der wird wissen, dass ich differentielle Genexpression als Folge einer Kaskade von hormonellen Signalen interpretiere, an deren Anfang Formatierungshormone stehen. Die nächste entwicklungsabhängige Stufe der Hormonkaskade bilden die von mir primäre Transkriptionshormone genannten Faktoren und die dritte und letzte schliesslich die sekundären Transkriptionshormone, die ich auch verschiedentlich als processing- und splicing-Hormone oder -Faktoren apostrophiert habe, weil sie mit eben diesem Mechanismus aus engste verknüpft sind. Während Formatierungshormone und primäre Transkriptionshormone etwa vom 5. Tag der Ontogenese an in die Regulation der Genexpression eingreifen, ist die Aktivität der sekundären Transkriptionshormone auf einen Zeitraum der Ontogenese beschränkt, der etwa mit dem 110. Tag beginnt. Dies ist gleichzeitig der Zeitpunkt, an dem die differentielle Genexpression ihren Anfang nimmt, der Zeitpunkt also, von dem an Primärtrankripte zu differenten messenger-RNAs verarbeitet, gespleisst werden.
 Das durch das ALU-Gen23 kodierte TAP mit 64 aa weist insgesamt fünf verschiedene Bindestellen für aktiverte HRPKS aus einem sekundären Transkriptionshormon und einem Rezeptorprotein auf. Das durch das ALU-Gen23 kodierte TAP mit 64 aa weist insgesamt fünf verschiedene Bindestellen für aktiverte HRPKS aus einem sekundären Transkriptionshormon und einem Rezeptorprotein auf.
Auch TAPs offerieren Bindestellen für Hormone, allerdings nicht für primäre Transkriptionshormone, sondern für die letztgenannten sekundären Transkriptions- oder Processing-Hormone, die von den erstgenannten aktiviert werden oder von ihnen abhängig sind. Während das SNIRP3 also durch Bindung eines TSH-HRPK an eine spezifische TSH-Bindestelle aktiviert wird, erfolgt die Aktivierung des zeitgleich transkribierten und translatierten TAP64 durch einen Hormon-Rezeptorproteinkomplex, dessen hormonelle Komponente von eben diesem TSH induziert wurde. Im konkreten Fall ist diese hormononelle TSH-abhängige Komponente das Hormon y-Lipotropin.
 |
Das TAP64 ist mit den gleichen Nukleosomengrössen korreliert, wie sein „kleiner Bruder„ (oder seine kleine Schwester), das SNIRP3 – die Expression von TAP64 ist über die ALU-Gene 22, 23 und 24 an die des SNIRP3 gekoppelt. Auch für die anderen in diesem Cluster kodierten SNIRPs und TAPs ergeben sich jeweils spezifische Kopplungsgruppen, deren Zusammenstellung sich aus diesem Chart ablesen lässt.
Das TAP64 weist fünf verschiedene Bindestellen für sekundäre Transkriptionshormone auf, die sämtlich von einem Formatierungshormon, bzw. einem primären Transkriptionshormon abhängig sind. Insgesamt weisen die im humanen Genom kodierten TAPs 52 verschiedene Bindestellen für sekundäre Transkriptionshormone auf – diese Zahl korrespondiert zum einen mit der Anzahl von Schlüsseln, die von der CTD209 der RNA-Polymerase II kodiert werden und zum anderen mit der Anzahl der von mir postulierten sekundären Transkriptionshormone. Darauf komme ich bei der Besprechung des Transkriptionskomplexes noch einmal zurück.
Wie und wann werden ALU-Gene transkribiert?
Da die durch ALU-Gene kodierten SNIRPs und TAPs ebenso wie die UsnRNAs für die Expression des metabolen Gens benötigt werden, das sich mit ihnen zusammen in der Subdomäne befindet, müssen sie in der Interphase zeitlich vor den metabolen Genen transkribiert werden. Die zeitlichen Abläufe dieser Prozesse werden unter anderem Gegenstand der folgenden Kapitel sein.
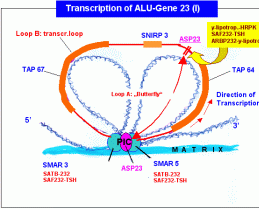 Die Transkription eines ALU-Gens wird durch einen Antisense-Promotor aktiviert, an den Faktoren gebunden haben, die durch die Hormonkaskade definiert sind. Dies sind ein zellspezifischer HRPK, ein zellspezifisches SAF- und ein zellspezifisches ARBP-Protein. Nach Bindung des Promotors an die SMARs und die Matrix und Bildung des PIC mit der RNAPII löst sich der Promotor wieder von der Matrix und das Gen wird ähnlich wie ein “normales” metaboles Gen transkribiert. Die Transkription eines ALU-Gens wird durch einen Antisense-Promotor aktiviert, an den Faktoren gebunden haben, die durch die Hormonkaskade definiert sind. Dies sind ein zellspezifischer HRPK, ein zellspezifisches SAF- und ein zellspezifisches ARBP-Protein. Nach Bindung des Promotors an die SMARs und die Matrix und Bildung des PIC mit der RNAPII löst sich der Promotor wieder von der Matrix und das Gen wird ähnlich wie ein “normales” metaboles Gen transkribiert.
Der eigentliche Transkriptionsprozess entspricht im Prinzip dem der REMA-Gene, denen sie in der Organisation in definierte Transkriptionseinheiten durch SMARS der Klasse II und NuMA-Proteine ja auch gleichen. Allerdings mit einigen Abweichungen.
Die Transkription eines ALU-Gens beginnt mit der Bindung spezifischer Architekturproteine an zwei SMARs, die Bindestellen für diese Proteine enthalten. Dies sind naturgemäss die gleichen Proteine, die auch bei der Transkription der UsnRNA-Gene die Funktion haben, die SMAR II an die Kernmatrix zu binden, also hormon-korrelierte Proteine der SATB-, SAF-, ARBP- und HMGB-Gruppe. Die stromab gelegenen SMAR II bindet wahrscheinlich zuerst an die Matrix, dann bindet die stromauf gelegene SMAR II an die gleiche Stelle – die beiden SMARs der gleichen Klasse bilden also einen Komplex. Dieser Prozess könnte durch das sogenannte mass-binding katalysiert werden, das für SAF-Proteine beschrieben wurde.
Zeitgleich oder anschliessend wird der antisense-Promotor (der durch die Formatierung der Domäne mit einer bestimmten NG determiniert ist) mit Hilfe eines zellspezifischen Hormon-Rezeptorproteinkomplexes und eines Proteins der ARBP-Familie an die beiden SMARs und damit auch an die Matrix gebunden. Dadurch entsteht zunächst die in der Skizze gezeigte Struktur, die einem Schmetterlingsflügel ähnlich sieht. Jetzt binden TFIID, TFIIB, die RNAPII und die restlichen GTFs - eine Helikase und eine Topoisomerase vervollständigen mit weiteren Proteinen den Transkriptionskomplex, der etwa 30 verschiedene Proteine umfassen dürfte. Eine spezifische Modifikation des ARBP232-y-Lipotropin-Proteins führt wahrscheinlich zur Lösung des Promotors von Matrix und Transkriptosom. Dadurch formiert sich eine Transkriptionsschleife (loop B), die im konkreten Fall von der SMAR3 bis zur SMAR5 reicht und jetzt das ALU-Gen mit seinen drei Exons/ORFs umfasst.
Loop B wird transkribiert (antisense-Transkription!). Die Transkription beginnt an einem definierten Punkt der SMAR5 und endet an einem definierten Punkt der SMAR3. Die mRNA wird mit einem cap versehen, polyadenyliert und nach Transport ins Zytoplasma dort translatiert. Spezifische Restriktionsenzyme (ALU-Gene!) zerlegen das Translationsprodukt anschliessend in einzelne Proteine, die in den Zellkern zurücktransportiert werden, wo sie für die Transkription des TPM1-Gens benötigt werden.
|